全載玻片成像( WSI ),即使用全載玻片掃描儀對載玻片上的組織進行數字化,正在醫療保健領域獲得廣泛關注。 WSI 使組織病理學、免疫組織化學和細胞學方面的臨床醫生能夠:
- 使用計算方法解釋圖像
- 將深度學習應用于數字圖像以提高臨床分析的準確性和再現性
- 提供有關患者數據的新見解
這篇文章解釋了 GPU 加速工具包如何提高輸入/輸出( I / O )性能和圖像處理任務。更具體地說,它詳細介紹了如何:
使用加速組織病理學技術節省時間對于快速識別和治療疾病和疾病至關重要。
WSI I / O 和圖像處理面臨的挑戰
將深度學習融入到整個幻燈片圖像的處理中需要的不僅僅是訓練和測試模型。使用深度學習的圖像分析需要大量的預處理和后處理來改善解釋和預測。整個幻燈片圖像必須準備好用于建模,并且生成的預測需要額外的解釋處理。示例包括偽影檢測、顏色歸一化、圖像二次采樣和去除錯誤預測。
此外,整個幻燈片圖像的大小通常非常大,分辨率高于 100000 x 100000 像素。這將強制平鋪圖像,這意味著在建模中使用來自整個幻燈片圖像的一系列子采樣。將這些平鋪圖像從磁盤加載到內存中,然后處理平鋪圖像可能非常耗時。
加速 WSI I / O 和圖像處理的工具
本文中介紹的用例使用 GPU 加速工具進行基準測試,具體如下。
cuCIM 公司
cuCIM ( C 計算 U 統一設備架構 C lara IM age ) 是一個用于多維圖像的開源加速計算機視覺和圖像處理軟件庫。用例包括生物醫學、地理空間、材料和生命科學以及遙感。 cuCIM 庫在許可證( Apache 2.0 )下公開可用,歡迎社區貢獻。
Magnum IO GPU 直接存儲
Magnum IO GPUDirect Storage (GDS) 提供存儲 I / O 加速,這是 Magnum IO 庫的一部分,用于并行、異步和智能數據中心 I / O 。 GDS 為 GPU 存儲器和存儲器之間的直接存儲器訪問( DMA )傳輸提供了直接數據路徑,從而避免了通過 CPU 的緩沖區反彈。此直接路徑增加了系統帶寬,減少了延遲,并減少了 CPU 的使用負載。
總體而言, GDS 具有以下優勢:
- 增加帶寬,減少延遲,并減少數據傳輸的 CPU 和 GPU 負載
- 減少了性能影響和對 CPU 處理存儲數據傳輸的依賴
- 對于完全遷移到 GPU 的計算管道,在計算優勢之上充當力倍增器
- 支持與其他基于開放源代碼的文件訪問的互操作性,通過使用傳統的文件 I / O (然后由使用 cuFile API 的程序訪問),可以將數據傳輸到設備或從設備傳輸數據
GDS 可以通過 kvikIO RAPIDS 包從 Python 訪問。 KvikIO 提供 Python 和 C ++ API ,能夠在 GDS 支持下執行讀或寫操作。(當 GDS 不可用時,返回到基本 POSIX 和cudaMemcpy操作。)
整個幻燈片圖像數據 I / O
當玻片在組織病理學中通過數字全玻片圖像掃描儀數字化時,高分辨率圖像將以多個放大倍數拍攝。 WSI 有一個金字塔形的數據結構,每個放大倍數的圖像形成一個 WSI 的“層”。這些圖像的最大放大倍數通常為 200 倍(使用放大倍數為 10 倍的 20 倍物鏡)或 400 倍(使用 10 倍的 40 倍物鏡)。
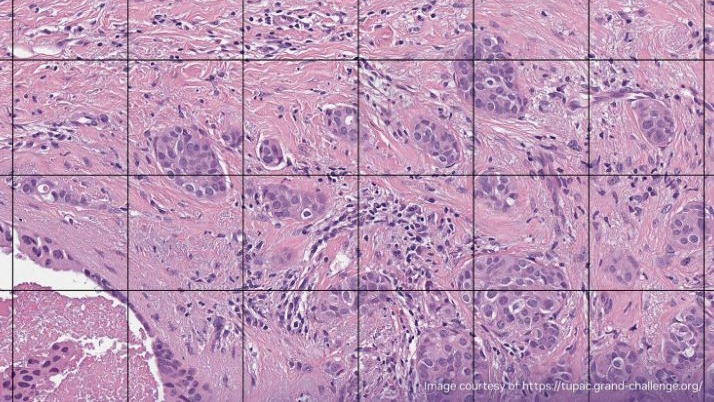
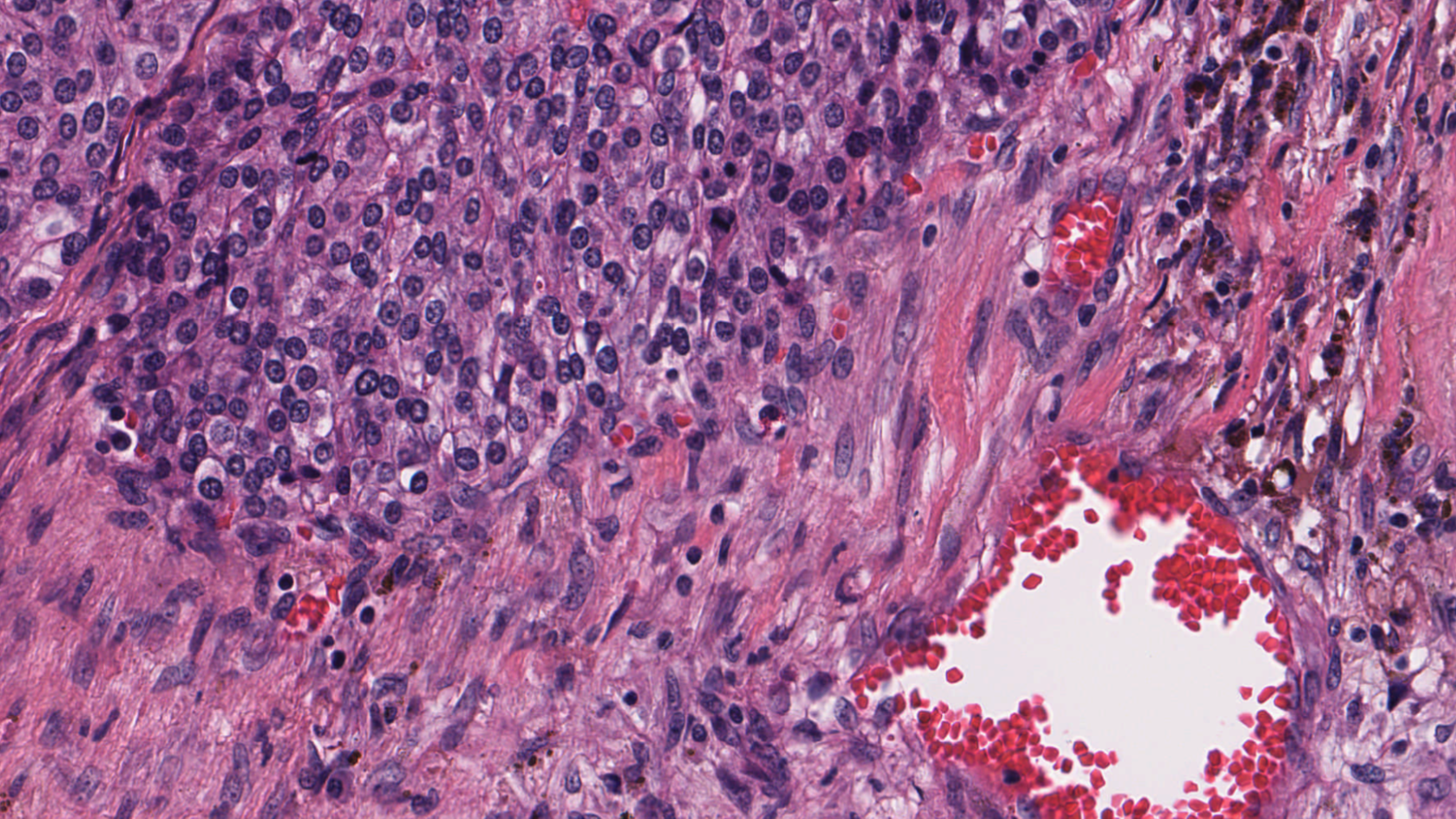
圖 1 和圖 2 顯示了乳腺癌研究的基準用例比較。圖 1 顯示了 H & E 染色數字病理切片的放大視圖,其中突出顯示了感興趣區域( ROI )。圖 2 顯示了與圖 1 中突出顯示的 ROI 相對應的高分辨率視圖。
為此用例選擇了具有以下特征的圖像數據集:
- 數字圖像中的層數為 XYZ
- 數字圖像的最高分辨率層,包含 2028 個大小( 512 、 512 )的紅、綠、藍( RGB )色塊
- 數字圖像的總大小為 XYZ

數字病理學用例
本節介紹了三種不同的數字病理學用例。
用例 1 :將 WSI 瓦片加載到 GPU 數組中
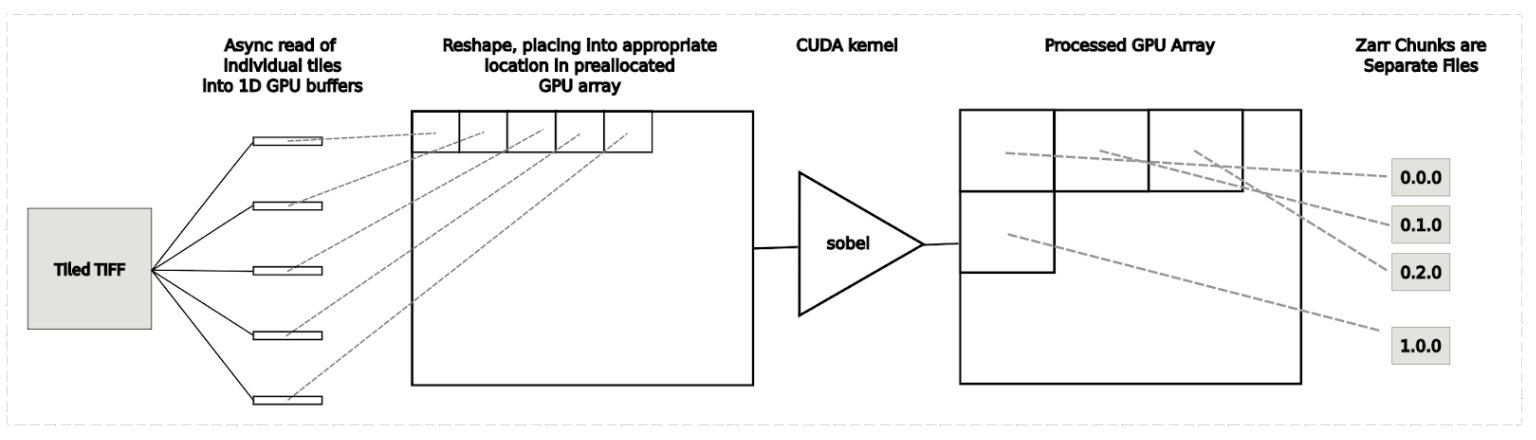
在這個圖像分析用例中,來自磁盤的 WSI 圖像中的每個單獨的區塊都被加載到具有和不具有 GDS 的 1D GPU 緩沖區中。目標 GPU 陣列已預先分配。在每次使用中,讀取區塊都會被重新整形并放置在 GPU 輸出陣列中的適當位置,如圖 3 所示。
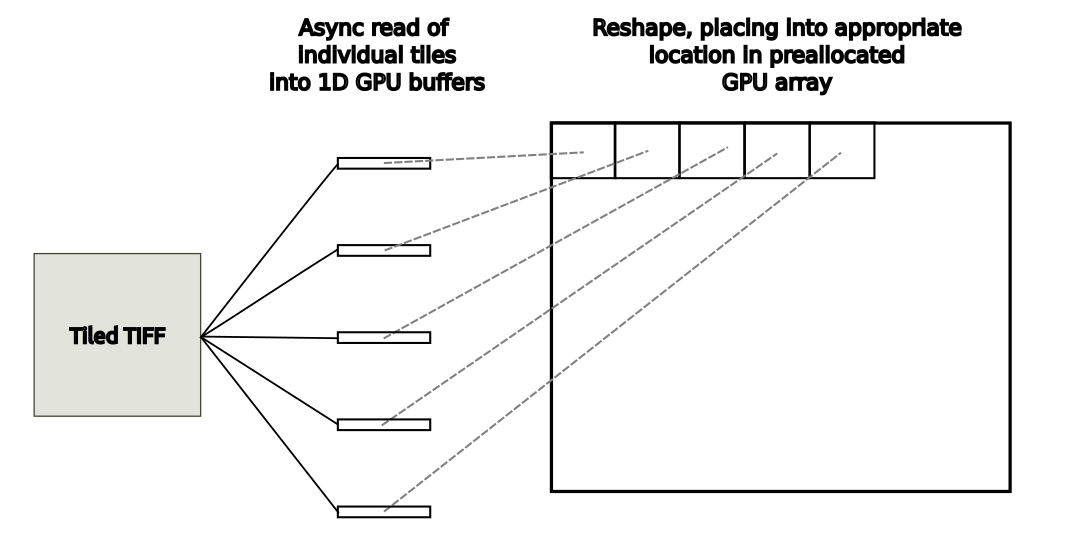
圖 4 所示的基準測試結果繪制了與tifffile.imread的使用相關的加速度,然后調用cupy.asarray將陣列從主機傳輸到 GPU 。如果沒有啟用 GDS , kivikIO 為單線程和并行讀取場景提供了 2.0 倍和 4.2 倍的加速。啟用 GDS 后,單線程情況下的加速從 2.0 倍提高到 2.7 倍,而并行讀取從 4.2 倍提高到 11.8 倍。
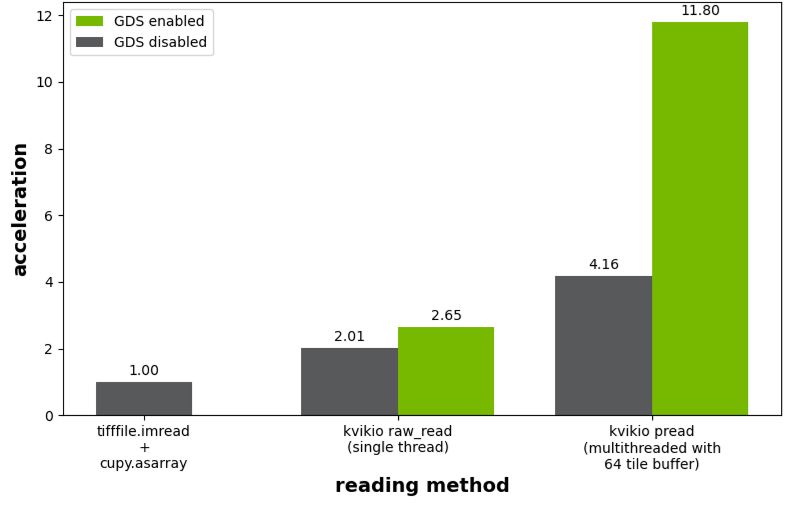
tifffile.imread和cupy.asarray的使用,平鋪 TIFF 圖像讀取的性能用例 2 :編寫未壓縮的 Zarr 文件
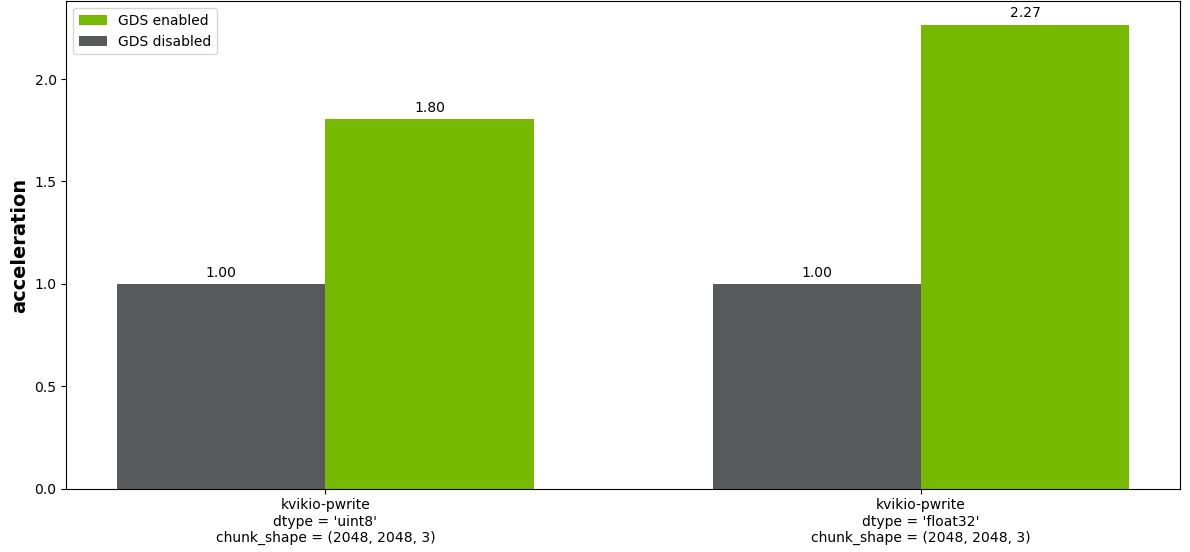
在此圖像分析用例中,從 GPU 內存中的 CuPy 陣列開始。例如,您可以從用例 1 的輸出開始。然后將此 GPU 數組平鋪寫入到單獨的 Zarr 文件中,包括 GDS 和不包括 GDS 。這涉及到將 6084MB 的數據以每個“塊”寫入獨立文件的格式寫入磁盤。各種塊形狀的文件大小如下:
- ( 256 、 256 、 3 )== 768 kB
- ( 512 、 512 、 3 )== 3 MB
- ( 1024 、 1024 、 3 )== 12 MB
- ( 2048 、 2048 、 3 )== 48 MB
圖 5 顯示了塊大小( 2048 、 2048 、 3 )的結果。對于除最小塊大小( 256 、 256 、 3 )之外的所有塊,啟用 GDS (未示出)具有顯著的好處。
用例 3 :平鋪圖像處理工作流
此圖像分析用例將加載、處理和保存整個幻燈片圖像合并到一個應用程序中。這個用例如圖 6 所示。步驟如下:
- 將單個平鋪加載到 1D GPU 緩沖區
- 將平鋪放置到預分配的 GPU 陣列的適當位置
- 使用基于 CUDA 的 Sobel 邊緣檢測內核的過程
- 將單個數據塊寫入 Zarr 文件到磁盤
一種方法是從用例 1 執行平鋪讀取,然后將 CUDA 內核應用于完整陣列,并從用例 2 執行平鋪寫入。這種方法具有相對較高的 GPU 內存開銷,因為完整圖像的兩個副本必須存儲在 GPU 內存中。在圖 8 所示的基準測試結果中,這種方法被標記為“多線程全局”
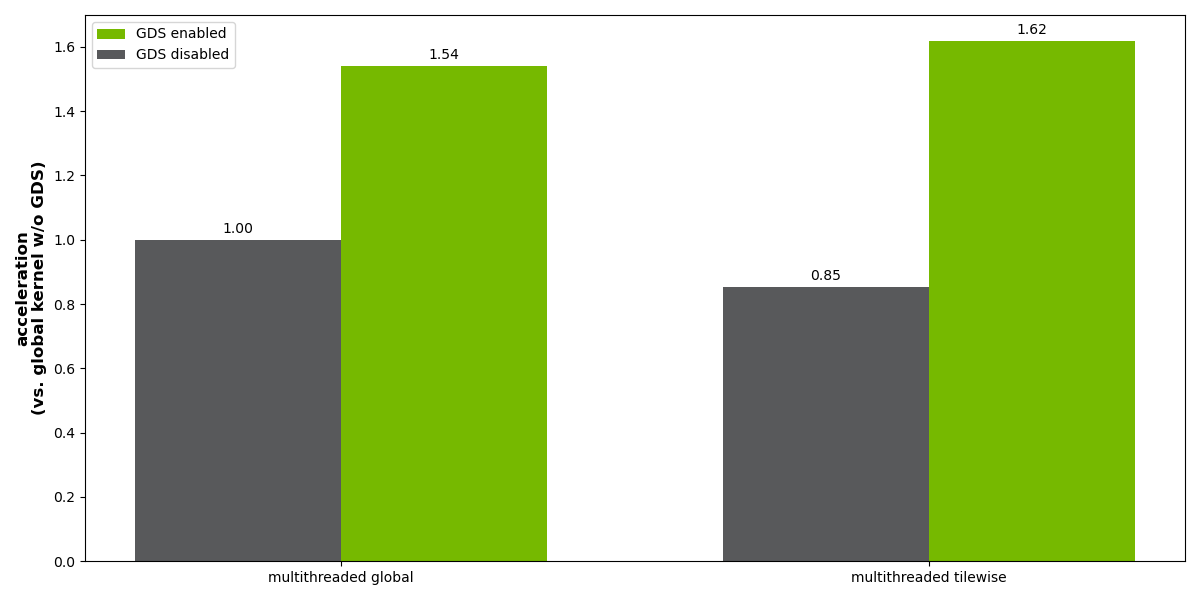
一種內存效率更高的方法是并行異步讀取、處理和寫入各個區塊。在這種情況下, GPU 內存需求大大減少,因為在任何一個時間只有一小部分圖像在 GPU 內存中。這種方法如圖 7 所示。在圖 8 所示的基準測試結果中,這種方法被標記為“多線程平鋪”
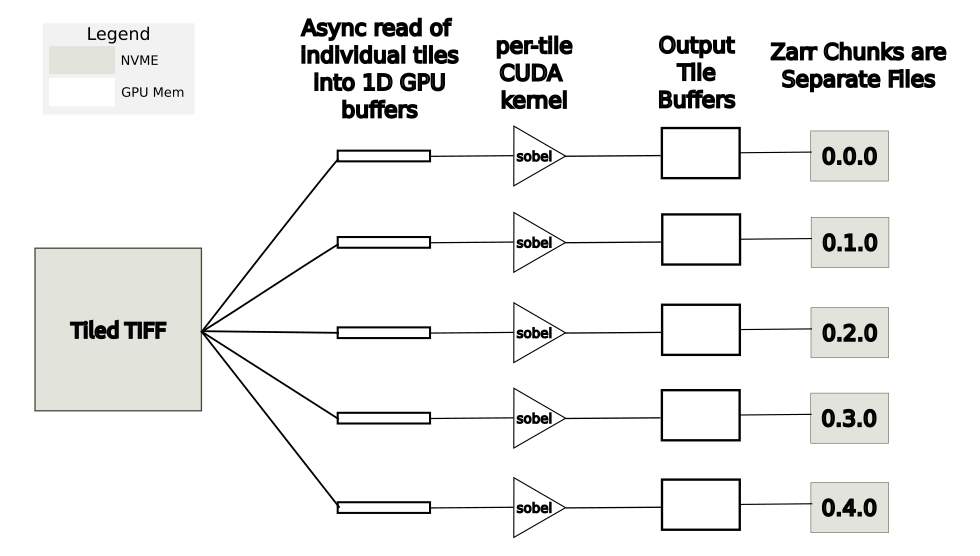
與禁用 GDS 的多線程全局方法相比,多線程平鋪方法的結果在性能方面是標準化的。在沒有 GDS 的情況下,執行異步平鋪處理會有 15% 的性能損失,但 GDS 運行時提高了約 2 倍,比使用 GDS 的全局方法略快。
平鋪 CUDA 處理方法的主要缺點是,由于平鋪處理,簡單實現目前無法處理潛在的邊界偽影。這與像素計算無關,如顏色空間轉換或數字病理學中從 RGB 到吸光度單位的轉換。
然而,對于涉及卷積的操作,使用單個瓦片的邊緣擴展而不是來自相鄰瓦片的實際數據可能會產生細微的偽影。為了處理類似的圖像分析用例,我們建議先保存到 Dask 數組,然后使用 map blocks 執行平鋪處理,這可以考慮這些邊界因素。
用于加速 WSI I / O 和圖像處理的示例腳本
用于基準測試的腳本可以從 cuCIM repository 的 examples / python / gds _ whole _ slide 文件夾中獲得。目前,這些圖像分析用例使用 RAPIDS kvikIO 進行 GDS 加速讀/寫操作。這些組織病理學演示僅供說明,尚未進行生產使用測試。未來,我們希望擴展 cuCIM API ,為執行這些類型的平鋪讀寫操作提供支持的方法。
總結:為什么加快 WSI I / O 至關重要
本文中介紹的圖像分析用例表明, NVIDIA GPUDirect Storage 在減少需要讀取和寫入平鋪數據集的各種高分辨率圖像的 I / O 時間方面具有顯著優勢。使用加速組織病理學技術可以節省時間,這對于快速識別和治療疾病和疾病至關重要。我們期待著將來在未壓縮的 TIFF 和 Zarr 數據上擴展這些初始結果,以包括壓縮數據。在此期間,我們邀請您使用 Conda 或 PyPI 下載 cuCIM 。 GDS 是 CUDA 11.8 的一部分。有關安裝要求,請參見 GDS 文檔 。
?