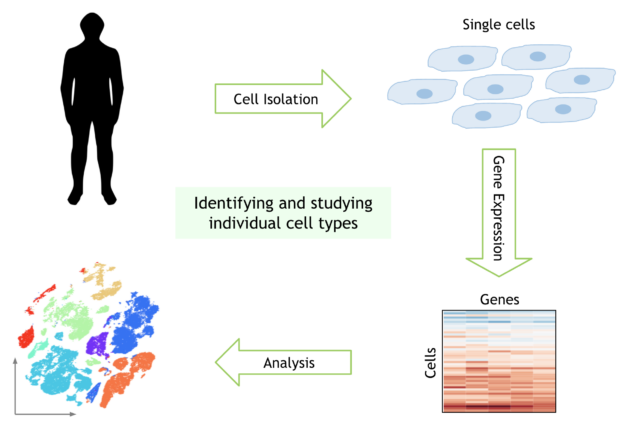
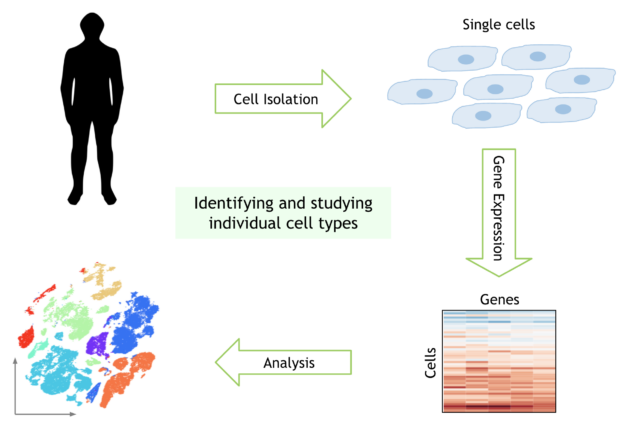
人體由近 40 萬億個細胞組成,有許多不同類型。實驗生物學的最新進展使探索單個細胞的遺傳物質成為可能。隨著單細胞基因組學這一新領域的誕生,科學家們現在可以探測人體內單個細胞的 DNA 和 RNA 。
單細胞基因組分析已經確定了人體內的新型細胞,發現了是什么使這些細胞彼此不同,以及不同類型的細胞如何對疾病或藥物作出反應。單細胞基因組學也被證明是當前 COVID-19 大流行的關鍵,它可以識別易受感染的細胞并揭示感染患者免疫系統的變化。
隨著最近的實驗對數百萬個細胞進行測序,單細胞數據的可用性和數據集的大小也在不斷增加。這種分析通常是探索性的,并從互動中得到進一步的好處——在更精細的尺度上識別不同類型的細胞,比較細胞類型并可視化它們之間的關系。當前的工作流仍然非常緩慢,這使得它們對于研究所需的交互分析來說是不可能的。
RAPIDS :用 GPUs 加速數據科學
RAPIDS 是一套開源庫,通過 GPU 加速的力量,可以加速端到端的數據科學工作流程。 RAPIDS 使得使用類似于 NumPy 、 pandas 和 scikit learn 的 Python api 對大型數據集執行交互式數據分析成為可能。
考慮執行單單元分析的典型工作流。這從一個矩陣開始,這個矩陣映射每個細胞中遇到的每個基因的數量。對數據進行預處理,濾除噪聲,然后對數據進行歸一化處理,得到每個細胞中每個人類基因的活性。在這一步中,機器學習也常用于糾正數據收集中的工件。接下來,在聚類和可視化之前執行維數縮減,以識別具有相似遺傳活動的細胞簇。最后,你比較這些細胞群的遺傳活動,以了解為什么不同類型的細胞表現和反應不同。
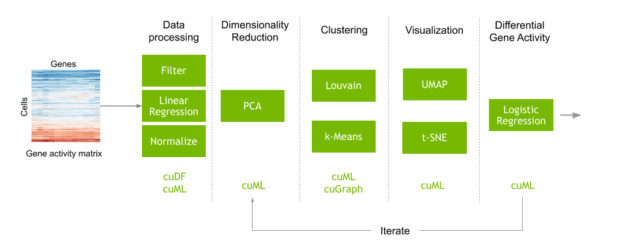
我們在 clara-parabricks/rapids-single-cell-examples GitHub repo 中發布了這個精確工作流的 GPU – 加速版本。 repo 包含一個示例 notebook ,它使用 RAPIDS 和 Scanpy 分析 70000 個人體肺細胞的數據集,以識別對 COVID-19 敏感的細胞。 Scanpy 是一個用于分析單細胞基因表達數據的工具包,提供了使用 RAPIDS 加速特定命令的選項。我們在回購中也有一個筆記本的 CPU 版本 以供比較。
例如,運行 UMAP 以使用 RAPIDS 可視化近 70000 個單元格需要以下命令:
sc.tl.umap(adata, min_dist=umap_min_dist, spread=umap_spread, method='rapids')
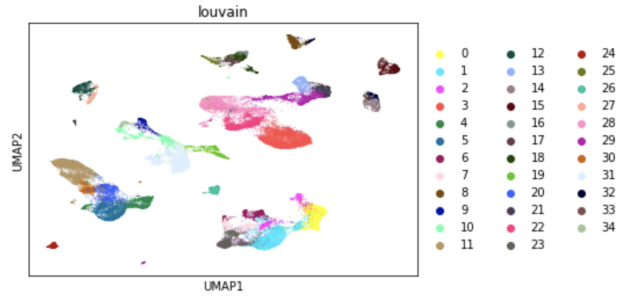
使用 RAPIDS 生成這個 UMAP 可視化需要 1 秒,而在 CPU 上則需要 80 秒。事實上, RAPIDS 可以加速整個單單元分析工作流程,甚至可以在大型數據集上進行交互式探索性數據分析。
| Instance | m5a.12xlarge | p3.2xlarge | Acceleration Factor |
| CPU/GPU type | Intel Xeon Platinum 8000, 48 vCPUs | V100-16GB | |
| Preprocessing | 311 | 84 | 4 |
| PCA | 18 | 3.4 | 5 |
| t-SNE | 208 | 2.2 | 95 |
| k-Means clustering | 31 | 0.4 | 78 |
| KNN | 25 | 6.1 | 4 |
| UMAP | 80 | 1 | 80 |
| Louvain clustering | 17 | 0.3 | 57 |
| Differential Gene Expression | 54 | 10.8 | 5 |
| End-to-end | 787 (13 Min) | 134 (2 Min) | 6 |
| Instance Price/hr ($) | 2.064 | 3.06 | ? |
| Total Run Cost ($) | 0.451 | 0.114 | 4 |
在 11 分鐘內分析一百萬個細胞
我們將我們的 RAPIDS 分析工作流程應用于現有最大的單細胞數據集之一, 100 萬個小鼠腦細胞通過 10 倍基因組學測序。有關詳細信息,請參閱 1M_brain_gpu_analysis_uvm.ipynb Jupyter 筆記本。
有了如此大的數據量,對 CPU 的分析變得不切實際地慢了下來;我們的端到端工作流在 awsm5a CPU 實例上運行了 3 個多小時。這使得交互式分析幾乎不可能。另一方面,我們在這個更大的數據集上觀察到了更高的 GPU 加速,并且能夠在一個 GPU 上分析整個數據集。在 AWS 上運行 RAPIDS 分析也比 CPU 版本便宜 3 倍!
| AWS Instance | m5a.12xlarge | p3.8xlarge | Acceleration Factor |
| CPU/GPU type | Intel Xeon Platinum 8000, 48 vCPUs | V100-16GB | |
| Preprocessing | 4033 | 323 | 12.5 |
| PCA | 34 | 20.6 | 1.7 |
| t-SNE | 5417 | 41 | 132.1 |
| k-Means clustering | 106 | 2.1 | 50.5 |
| KNN | 585 | 53.4 | 11.0 |
| UMAP | 1751 | 20.3 | 86.3 |
| Louvain clustering | 597 | 2.5 | 238.8 |
| End-to-end | 13002 | 672.7 | 19.3 |
| Instance Price/hr ($) | 2.064 | 12.24 | ? |
| Total Run Cost ($) | 7.455 | 2.287 | 3.3 |
用于交互式單細胞分析的 GPU 功能單元瀏覽器
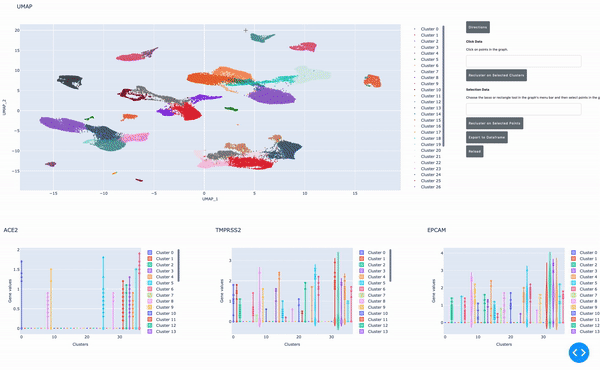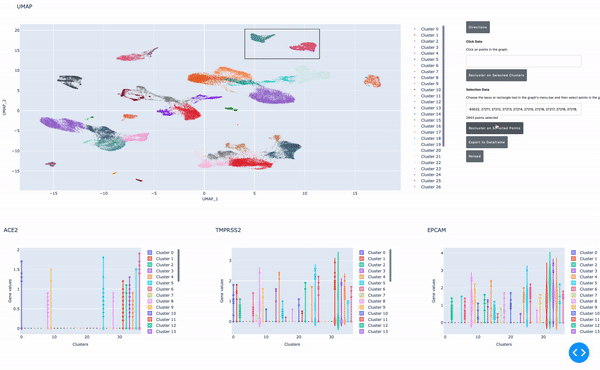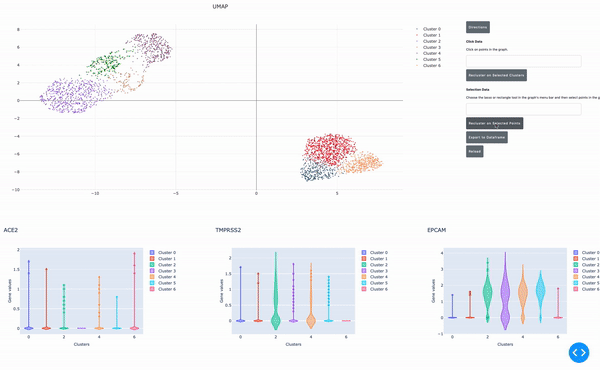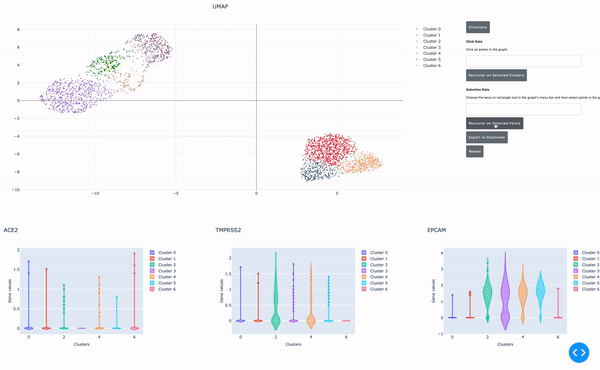
如前所述, RAPIDS 的數據分析速度使研究人員能夠實時交互式地分析數據。我們開發了一個在 Jupyter 筆記本 中運行的、支持 GPU 的交互式小區瀏覽器,使這一過程更加簡單。在這個單元格瀏覽器中,您可以可視化數據集中的所有單元格,并通過點擊方法對數據執行聚類分析。使用 RAPIDS ,這些步驟可以實時運行。
在這篇文章中,我將向您展示如何輕松地選擇一組細胞,并執行 UMAP 和 Louvain 聚類來識別這種細胞類型中的子種群。
結論
在這篇文章中,您看到了使用 RAPIDS 加速 GPUs 上的單細胞基因組分析是多么容易。使用 RAPIDS ,可以方便地實時交互地探索數據,對不同尺度的單元進行聚類,以及對具有不同參數的大型數據集進行重新分析。所有這些都有助于更快的科學發現。
除了涵蓋的 API 之外, RAPIDS 還有一個大型的其他算法庫,您會發現這些算法在您的工作中很有用。有關更多信息,請參閱 clara-parabricks/rapids-single-cell-examples GitHub repo 以了解此工作以及 RAPIDS 。
?