單細胞基因組學研究繼續推進疾病預防藥物的發現。例如,它在為當前的新冠肺炎大流行開發治療、識別易受感染的細胞以及揭示受感染患者免疫系統的變化方面起著關鍵作用。然而,隨著大規模單細胞數據集可用性的不斷提高,計算效率的低下明顯影響了科學研究的速度。將這些計算瓶頸轉移到 GPU 已經證明了有趣的結果。
在最近的一篇博客文章中, NVIDIA 對 100 萬個小鼠腦細胞進行了基準分析,這些腦細胞通過 10 倍基因組學測序。結果表明,在 GCP CPU 實例上運行端到端工作流需要三個多小時,而在單個 NVIDIA V100 GPU 上處理整個數據集只需 11 分鐘。此外,在 GCP GPU 實例上運行 RAPIDS 分析的成本也比 CPU 版本低 3 倍。此處閱讀博客。
按照Jupyter 筆記本對該數據集進行 RAPIDS 分析。要運行筆記本,文件rapids_scanpy_funcs.py和utils.py必須與筆記本位于同一文件夾中。我們提供了第二個筆記本,其中包含 CPU 版本的分析here。在與 Google Dataproc 團隊的合作下,我們構建了一個入門指南,以幫助開發人員快速運行這個轉錄組學用例。最后,看看這個 NVIDIA 和谷歌云共同撰寫博客文章,它展示了工作的影響。
對 GPU 進行單細胞 RNA 分析
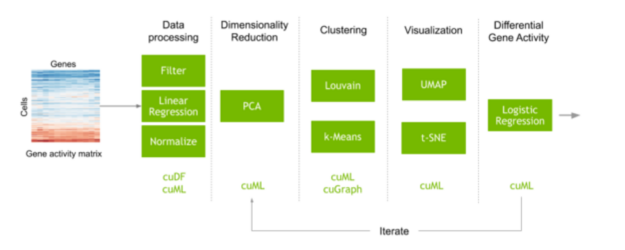
執行單細胞分析的典型工作流程通常從一個矩陣開始,該矩陣映射每個細胞中測量的每個基因腳本的計數。執行預處理步驟以濾除噪聲,并對數據進行歸一化以獲得在數據集的每個單獨單元中測量的每個基因的表達。在這一步中,機器學習也常用于糾正數據收集中不需要的偽影。基因的數量通常相當大,這會產生許多不同的變異,并在計算細胞之間的相似性時增加很多噪音。在識別和可視化具有相似基因表達的細胞簇之前,特征選擇和降維可以減少這種噪聲。這些細胞簇的轉錄表達也可以進行比較,以了解為什么不同類型的細胞行為和反應不同。
該分析證明了使用RAPIDS加速使用單個 GPU 分析 100 萬個細胞的單細胞 RNA 序列數據。然而,實驗只處理了前 100 萬個細胞,而不是整個 130 萬個細胞。因此,在單細胞 RNA 數據的工作流中處理所有 130 萬個細胞的時間幾乎是單個 V100 GPU 的兩倍。另一方面,相同的工作流在單個 NVIDIA A100 40GB GPU 上只需 11 分鐘。不幸的是, V100 的性能下降了近 2 倍,主要原因是 GPU 的內存被超額訂閱,從而在需要時溢出到主機內存。在下一節中,我們將更詳細地介紹這種行為,但需要明確的是, GPU 的內存是擴展的限制因素。因此,更快地處理更大的工作負載需要更強大的 GPU 服務器,如 A100 或/或將處理分散到多個 GPU 服務器上。
將預處理擴展到多個 GPU 的好處
當工作流的內存使用量超過單個 GPU 的容量時,統一虛擬內存( UVM )可用于超額訂閱 GPU ,并自動溢出到主內存。這種方法在探索性數據分析過程中是有利的,因為適度的超額訂閱率可以消除在 GPU 內存不足時重新運行工作流的需要。
但是,嚴格依靠 UVM 將 GPU 的內存超額訂閱 2 倍或更多可能會導致性能不佳。更糟糕的是,當任何單個計算需要的內存超過 NVIDIA 上的可用內存時,它可能會導致執行無限期掛起。將計算擴展到多個 GPU 可以提高并行性并減少每個 GPU 上的內存占用。在某些情況下,它可以消除超額認購的需要。圖 2 表明,我們可以通過將預處理計算擴展到多個 GPU 來實現線性縮放,與單個 GPU V100 GPU 相比, 8 個 GPU s 會產生略微超過 8 倍的加速比。考慮到這一點,需要不到 2 分鐘才能將 130 萬個細胞和 18k 基因的數據集減少到約 129 萬個細胞和 8 GPU上 4k 個高度可變的基因。這超過了 8 . 55 倍的加速,因為單個 V100 需要 16 分鐘來運行相同的預處理步驟。
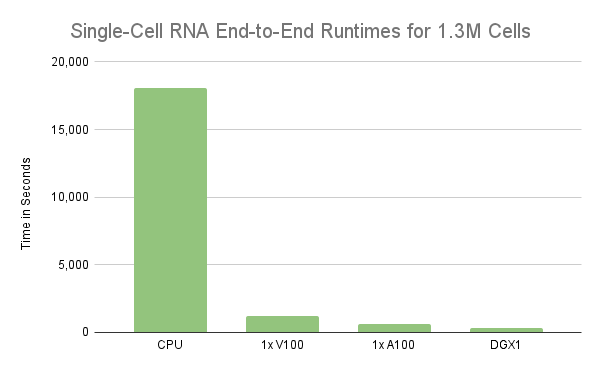
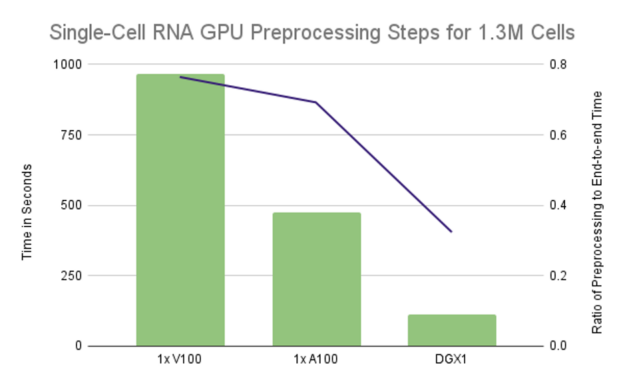
圖 3 :單個 GPU 配置的運行時主要由預處理步驟控制,在單個 V100 上占據 75% 的端到端運行時,在單個 A100 上占據 70% 的運行時。利用 DGX1 上的所有 GPU 將比率降低到略高于 32%.
使用 Dask 和 RAPIDS 將單細胞 RNA 筆記本擴展到多個 GPU
許多預處理步驟,如加載數據集、過濾嘈雜的轉錄本和細胞、將計數標準化為表達式以及特征選擇,本質上是并行的,每個 GPU 獨立負責其子集。糾正數據收集噪音影響的一個常見步驟是使用不需要的基因(如核糖體基因)的貢獻比例,并擬合許多小型線性回歸模型,數據集中每個轉錄本對應一個模型。由于轉錄本的數量通常可以達到數萬個,因此通常使用分散性或可變性的度量方法,只選擇幾千個最具代表性的基因。
Dask是一個優秀的庫,用于在一組工作進程上分發數據處理工作流。 RAPIDS 通過將每個工作進程映射到自己的 GPU ,使 Dask 也能夠使用 GPU s 。此外, Dask 提供了一個分布式陣列對象,非常類似于 NumPy 陣列的分布式版本(或CuPy,其 GPU 加速外觀相似),它允許用戶在多個 GPU 上,甚至跨多臺物理機器,分發上述預處理操作的步驟,操作和轉換數據的方式與 NumPy 或 CuPy 數組大致相同。
在預處理之后,我們還通過對數據子集進行訓練并分配推理來分配主成分分析( PCA )縮減步驟,通過僅將前 50 個主成分恢復到單個 GPU 來降低通信成本,用于剩余的聚類和可視化步驟。該數據集的 PCA 簡化單元矩陣僅為 260 MB ,允許在單個 GPU 上執行剩余的聚類和可視化步驟。使用這種設計,即使包含 500 萬個單元的數據集也只需要 1GB 內存。
結論
以我們計算工具的發展速度,我們可以假設數據處理量很快就會迎頭趕上,特別是對于單細胞分析工作負載,這迫使我們需要更高的擴展。同時,通過將聚類和可視化步驟分布在多個 RAPIDS 上,仍有機會進一步減少探索性數據分析過程的迭代次數。更快的迭代意味著更好的模型,縮短洞察時間,以及更明智的結果。除 T-SNE 外,多 GPU 工作流筆記本的所有集群和可視化步驟都可以通過 GPU cuML 和 cuGraph 分布在 GPU 上的 Dask 工作人員上。
?