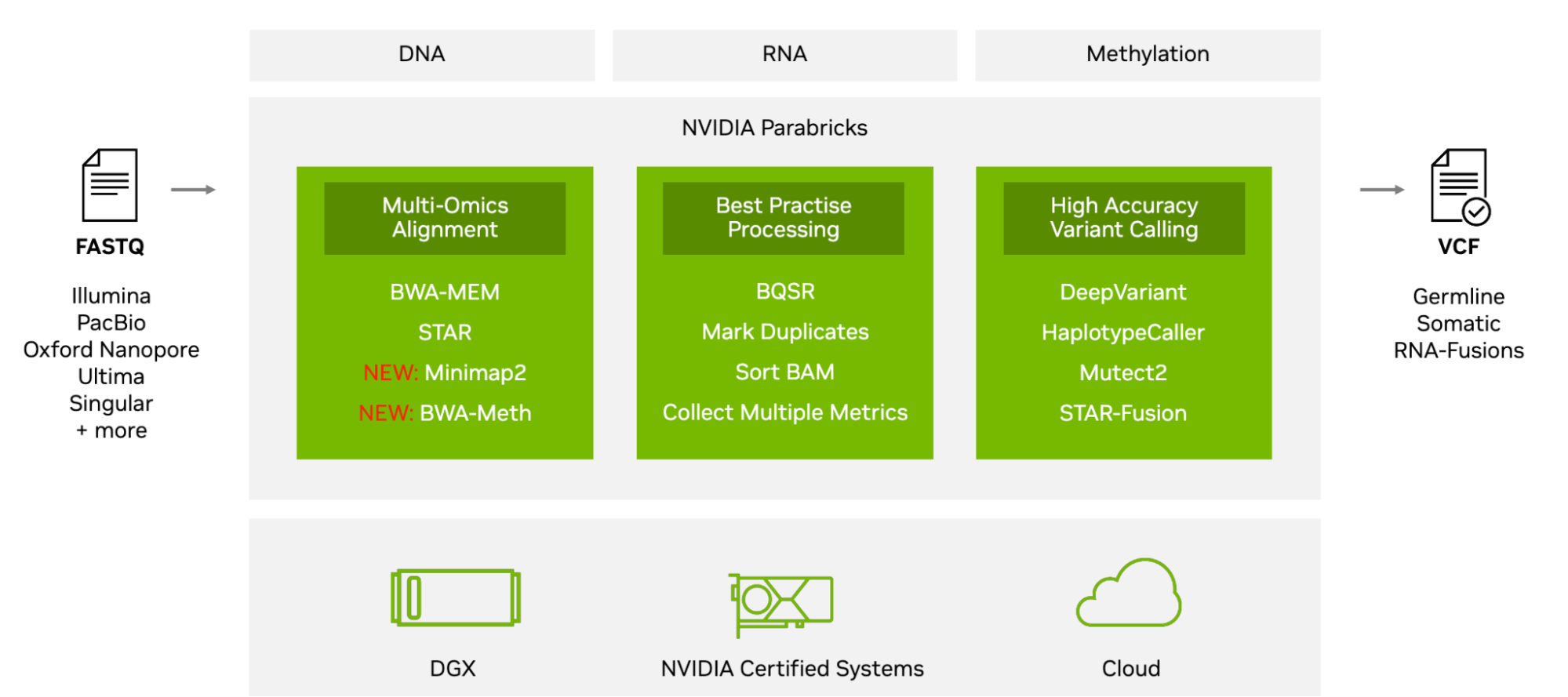
NVIDIA Parabricks v4.3 在 NVIDIA GTC 2024 上發布,引入了新的工具和工作流程,為多種組學數據類型帶來了加速和新的 AI 技術。除了分析 DNA 和 RNA,您現在還可以利用 GPU 和生成式 AI 的強大功能,以高速和高精度分析甲基化、單細胞和空間組學工作負載。
Parabricks v4.3 還進一步優化了生殖細胞工具,并支持最新的 GPU 架構,進一步縮短了分析時間。
新內容
- 全新的單細胞和空間組學數據工作流程,整合了整個 NVIDIA 的軟件 (包括成像、生成式 AI 和加速數據科學),可實現快速、高精度的分析。
- BWA-Meth 的加速版本,作為 Parabricks 中的一種新工具,可加速對 DNA 甲基化數據的亞半酸雙星測序比對。
- 進一步優化種系分析,使用 NVIDIA H100 動態編程核心將種系分析縮短到 10 分鐘以內,現已成為市場上最快的黃金標準工具 (BWA-MEM 和 GATK)
- 支持最新的 NVIDIA 數據中心 GPU,包括 NVIDIA Grace Hopper
- DeepVariant 到 v1.6 的升級版本,支持 NovaSeqX 數據
單細胞和空間組學
有一些技術使研究人員能夠在細胞層面理解組學,例如單細胞測序,并通過空間分辨率方法將這些數據放入組織環境中。這些技術作為一種為細胞和組織基礎的生物系統建模的方法越來越受歡迎。
這給行業帶來了一些新挑戰。
首先,單細胞和空間實驗所產生的數據規模正在飛速發展。單細胞圖譜項目已達到數百萬個細胞的數量級,而新一代空間組學儀器所產生的數據量約為 PB 級的成像數據。
其次,對此的分析需要更多的自動化。基于圖像的空間方法需要對細胞進行分割,并在多達數十萬個細胞中量化表達值。分割是一項并非易事的任務,使用許多方法 (包括人類在環) 在面對新的數據集或細胞類型時指導算法。
最后,生成的數據為獲得見解提供了全新的可能性,不僅提供了可用的表達數據,而且圖像細胞的形態也為研究人員提供了潛在的有用信息。
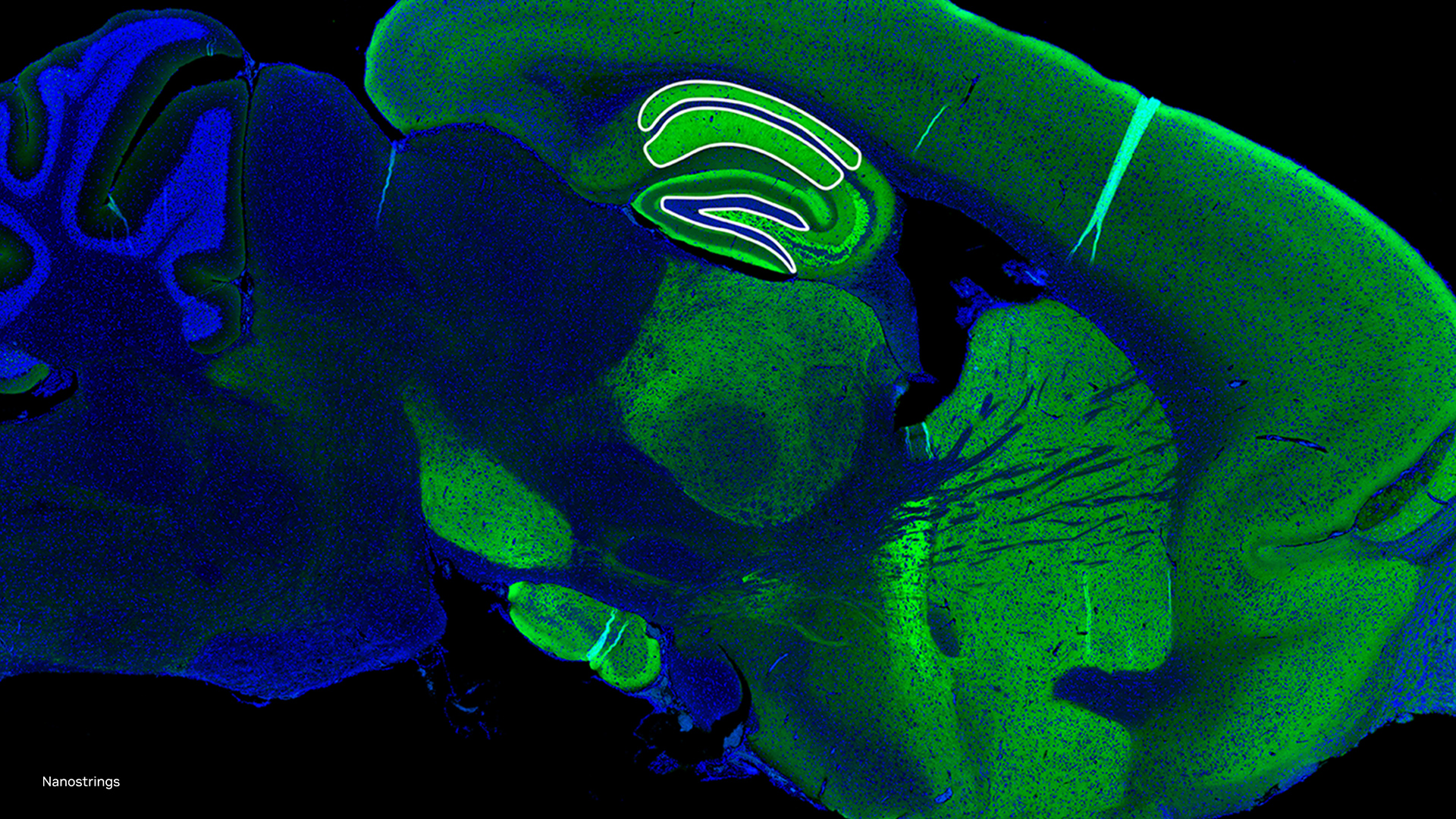
空間組學提供商,如 Nanostring,使用 NVIDIA GPU 并在 CosMx SMI 設備上加速計算,以應對這些挑戰。
“空間組學技術(CosMx 空間分子成像)現在可以對細胞和組織內的整個轉錄組進行成像,以前所未有的密度和規模(超過 150 TB/cm2)生成數據。這些數據將在轉變我們對健康和疾病的理解方面發揮關鍵作用,從根本上加速藥物研發和空間診斷,”NanoString 首席科學官兼研發高級副總裁 Joseph Beechem 博士說。
“事實上,探索這些圖像的真實信息內容需要生成式 AI.我們很高興繼續在數據到信息流程的各個層面上深化與 NVIDIA 的合作。我們邀請所有 AI/ML 社區加入我們,共同參與這場生命科學空間生物學革命。”
為此, NVIDIA Parabricks 現在包含單單元和空間組學參考工作流程 (圖 1),以加速計算瓶頸,提供更高水平的準確性,并啟用新的分析方法。
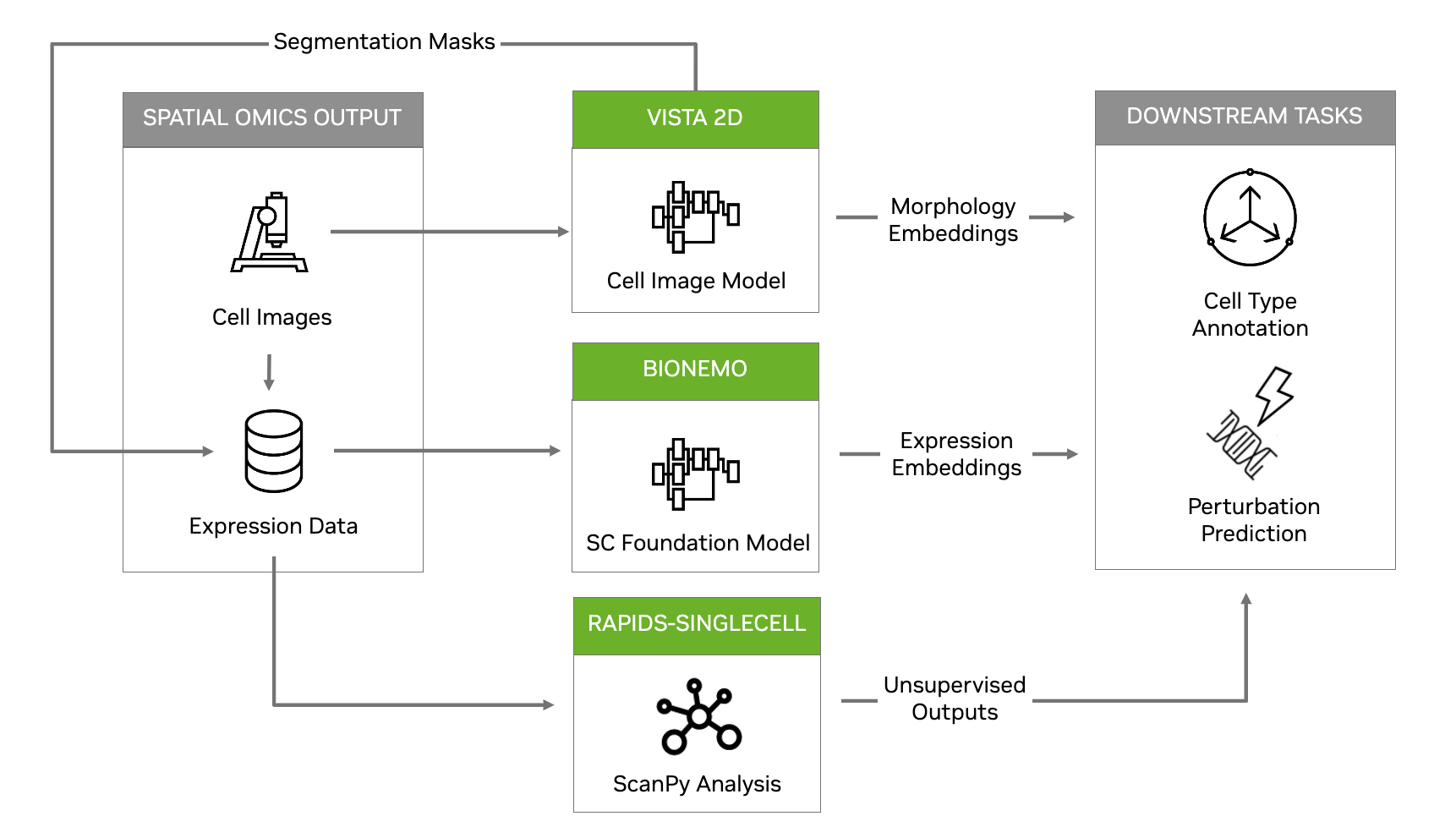
為了解決單單元和空間表達式輸出分析中的瓶頸問題, NVIDIA 最近與柏林的 Charit é 合作,將 NVIDIA RAPIDS 引入 scverse 生態系統,生成了一個新的 RAPIDS-SingleCell 庫。該庫主要用于加速 Scanpy 的嵌入式替代,在某些情況下,單個 A100 GPU 可實現高達 850 倍的加速。
現在, NVIDIA 將生成式 AI 引入單細胞和空間組學分析,新的細胞成像基礎模型能夠實現高精度分割 (對于將表達正確歸因于正確的細胞非常重要,會影響所有下游任務的準確性),并且能夠生成表示細胞形態的嵌入。
我們 NVIDIA BioNeMo 框架 還發布了用于構建和訓練單細胞 BERT 模型的功能,這些功能可以應用于單細胞表達數據的微擾預測等生成任務。
現在,所有這些內容都已整合到一個參考工作流程中,并被維護于在公共的? /clara-parabricks-workflows GitHub 庫中。
Parabricks v4.3 工具和基準測試
除了在單細胞和空間領域的開發之外,我們還發布了 NVIDIA Parabricks 4.3 版,進一步推進了 Parabricks 的使命,即加速所有測序儀和組學的比對和變異識別。
此版本進一步突破了測序分析的瓶頸,優化了以下行業信賴的工具:
- BWA-MEM
- GATK BQSR
- 標記重復項
- 單倍型識別程序
- DeepVariant
通過使用 NVIDIA H100 DPX 指令進行開發,此 Parabricks 版本縮短了端到端分析的運行時間,從而取得了突破性成果。
Oracle Cloud 的工程師已經在創紀錄的時間內成功運行了這一工作流程。“我們很高興 NVIDIA 從硬件和軟件的角度持續加速基因組分析,”Oracle 全球 AI 云總監 Dan Spellman 說,“通過使用最新版本的 Parabricks 和 H100,由 Ruzhu Chen 領導的 OCI 的基因組學工程將處理時間縮短到 10 分鐘以下。”
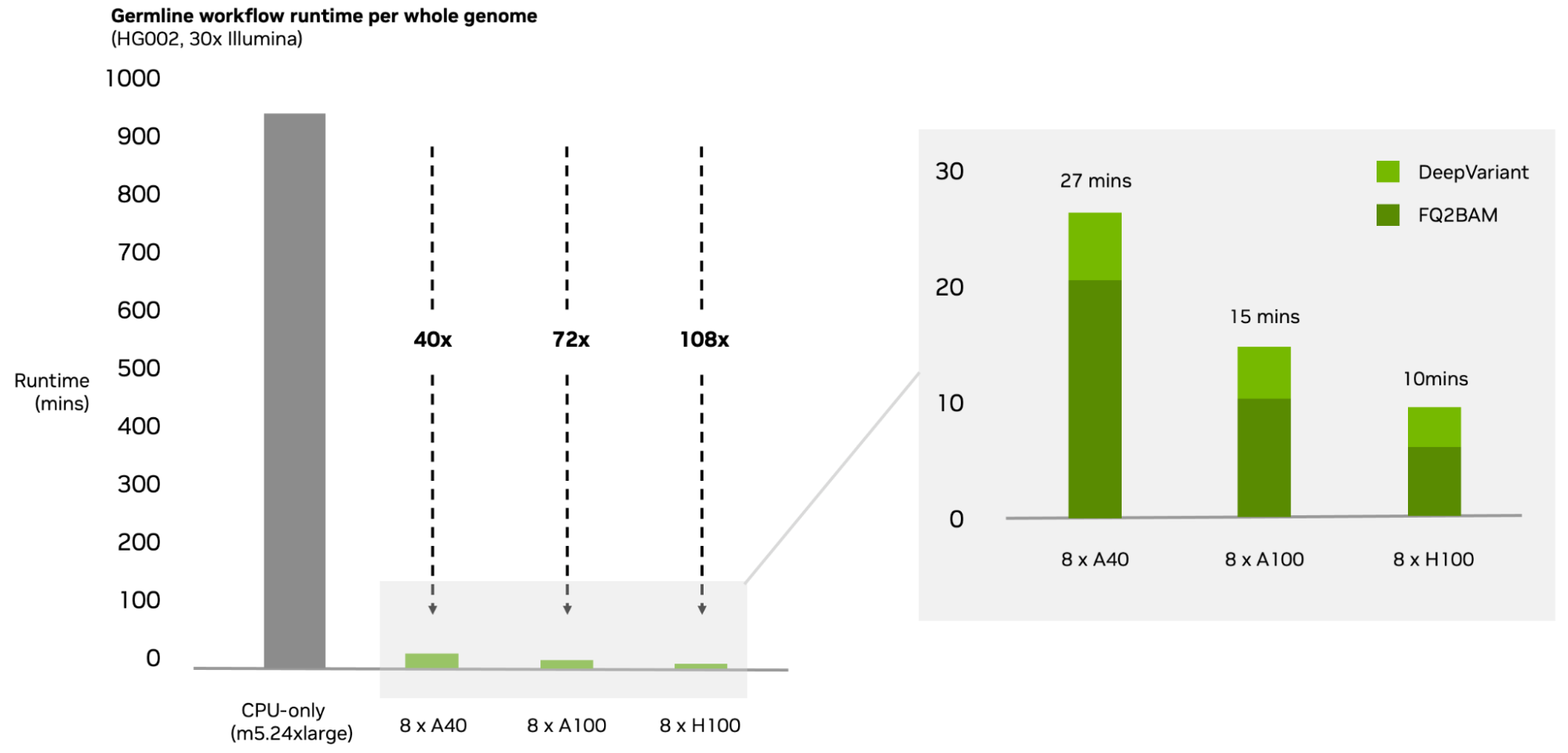
版本 4.3 中工具的進一步開發包括對 GPU 加速的 Minimap2 的運行時改進,用于對齊 PacBio 數據,以及名為 fq2bam_meth 的 BWA-Meth 的加速版本,用于進行甲基化數據比對。
DNA 甲基化是表觀基因組的關鍵組成部分,在調節不同組織中的基因表達方面發揮著重要作用。對甲基化和表觀基因組的研究表明,這些可能是疾病的致病因素,并且可以為心血管疾病測試或液體活檢等應用提供關鍵標記,以便早期檢測血液中的癌癥。
BWA-Meth 是一種用于準確比對雙亞述轉換 DNA 讀取的工具 (通過將所有非甲基化的細胞因子轉化為來揭示甲基化的過程)。與僅使用 CPU (c5.12 xlarge,48 個 CPU 線程) 相比,在 Parabricks 中加速 BWA-Meth 可在 NVIDIA DGX A100 (8 個 NVIDIA A100 GPU) 上實現高達 36 倍的加速,運行在全基因組雙亞述測序數據上。
Sequanta 是中國的一家多組學研究和臨床服務提供商,其研究人員使用 Parabricks BWA-Meth 已實現比其他比對方法快 21 倍的加速。他們現在能夠在 8 個 T4 GPU 上在短短 60 分鐘內對甲基化樣本進行比對,而其他方法需要 21 個小時。
了解詳情
NVIDIA Parabricks v4.3 提供加速的多組學分析,解決了 DNA、RNA、甲基化、單細胞和空間組學數據方面的挑戰。它為單細胞和空間組學提供了參考工作流程,加速了分析并實現了新方法。
通過優化和對行業信賴工具的支持,Parabricks 將運行時間縮短到 10 分鐘以下,打破了測序分析的瓶頸。
從 NGC 下載 Parabricks v4.3 容器 和 訪問 GitHub 上的參考工作流 以加速多組分析,并獲得對生物系統的更深入見解。開始使用 Parabricks 并發揮加速基因組分析的潛力。
?