快速且經濟高效的全基因組測序和分析可以迅速為患有罕見或未診斷疾病的危重患者提供答案。最近在加速臨床測序方面取得的進展,例如創造世界紀錄 用于快速診斷的 DNA 測序技術 ,使我們離在臨床環境中進行全基因組基因診斷又近了一步。
斯坦福大學醫學院( Stanford University School of Medicine )、NVIDIA ( NVIDIA )、谷歌( Google )、 UCSC 和牛津納米孔技術( Oxford Nanopore Technologies , ONT )領導的一個團隊最近使用這項技術來識別與疾病相關的基因變異,這些變異在短短 7 小時 18 分鐘內就得到了診斷,結果于 2022 年 1 月發表在 新英格蘭醫學雜志 上。
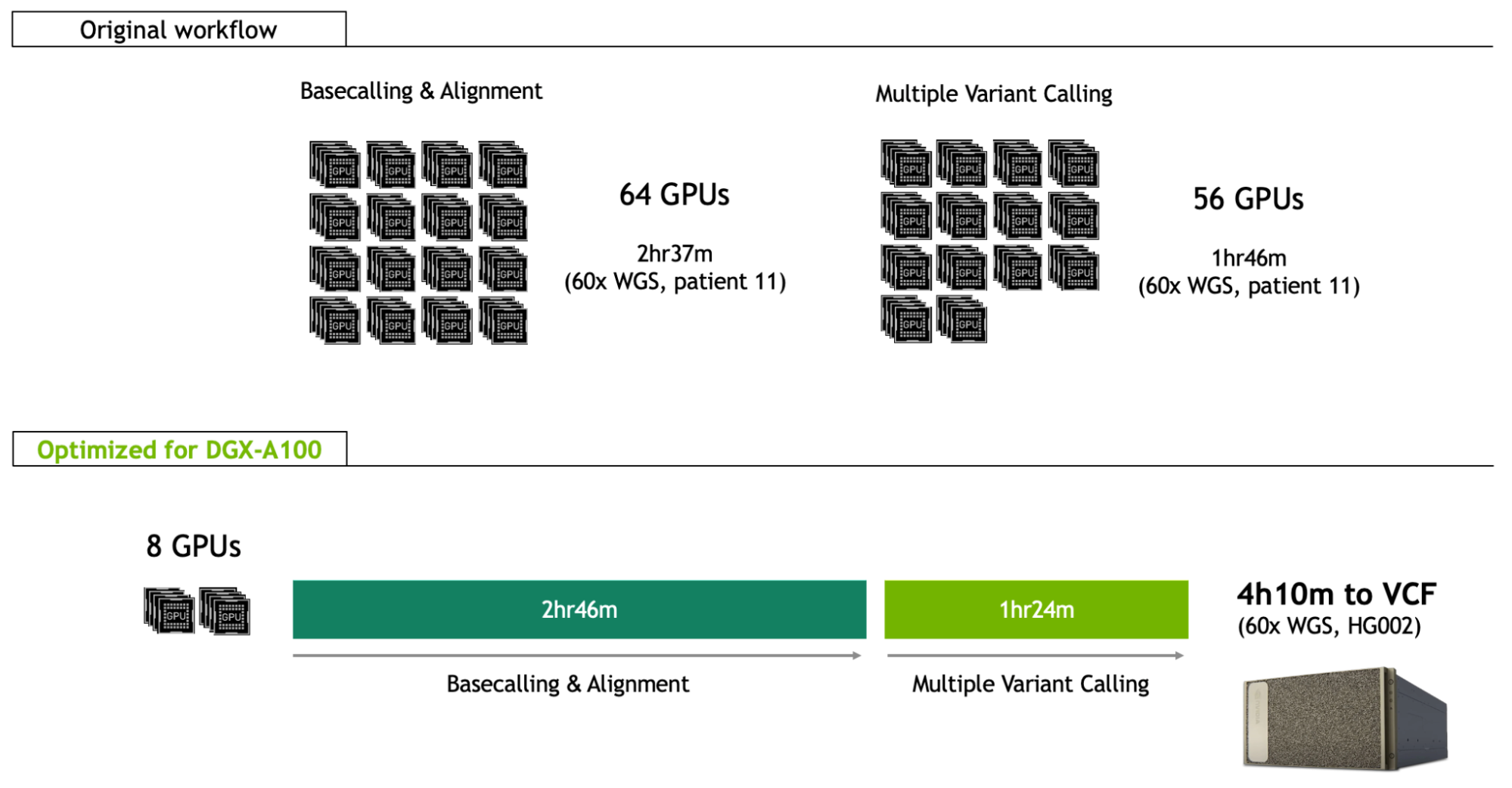
這一創紀錄的端到端基因組工作流程依賴于創新技術和高性能計算。它利用長閱讀納米孔測序技術更好地分析結構變體。這是在 48 個流動池中實現的,優化的方法使孔占有率達到 82% ,在短短幾個小時內快速生成 202 千兆堿基。對輸出的分析分布在一個谷歌云計算環境中,包括 16 個 4xV100 GPU 實例(總計 64 GPU 個)的基調用和對齊,以及 14 個 4xP100 GPU 實例(總計 56 GPU 個)的變體調用。
自一月 NEJM 發表以來,NVIDIA Clara 團隊一直在優化 DGX-A100 的全基因組工作流程,使臨床醫生和研究者能夠在八 A100 GPU 上部署與世界記錄方法相同的分析,而在 4H10M 中部署 60X 全基因組(圖 1 ;在 HG00 參考樣品上標明)。
這不僅可以在本地運行的單服務器( 8-GPU )框架中實現快速分析,還可以將每個樣本的成本降低三分之二,從 568 美元降至 183 美元。
基本呼叫和對齊
堿基調用是將原始儀器信號分類為基因組堿基 A 、 C 、 G 和 T 的過程。這是確保所有下游分析任務準確性的計算關鍵步驟。這也是一個重要的數據縮減步驟,將生成的數據縮減約 10 倍。
以每堿基 340 字節為單位,一個單一的 60 倍覆蓋率的整個基因組在原始信號中很容易達到數萬億字節,而在處理時則為數百千兆字節。因此,計算速度有利于與測序輸出速度相匹敵,這是非常重要的,通過 48 個流動單元的 128000 個孔,以每秒約 450 個堿基的速度進行。
ONT 的 PromethION P48 測序儀在 72 小時的運行中可以產生多達 10 個 Terabase ,相當于 96 個人類基因組(覆蓋率為 30 倍)。
這項工作所需的快速分類任務已經受益于深度學習創新和 GPU 加速。用于此目的的核心數據處理工具包 Guppy 使用遞歸神經網絡( RNN )進行基址調用,可以選擇更小(更快)或更大(更高精度)的遞歸層大小的三種不同架構。
BaseCall 中的主要計算瓶頸是 RNN 內核,它得益于 GPU 與 ONT 序列器的集成,例如桌面網格 Mk1 ,其中包括一個 V100 GPU 和手持 MinION Mk1C ,其中包括一個 Jetson 邊緣平臺。
比對是將合成的堿基 DNA 片段(現在是 As 、 Cs 、 Gs 和 Ts 的字符串形式)提取出來,并確定這些片段起源的基因組位置,通過大規模并行測序過程組裝完整基因組的過程。這基本上是從許多 100-100000 bp 長的讀取中重建全長基因組。就創造世界紀錄的樣本而言,總共有 1300 萬次閱讀。
在最初的世界記錄分析中, basecalling 和 alignment 分別在 Guppy 和 Minimap2 的不同實例上運行。通過將其遷移到單服務器 DGX-A100 解決方案,并使用 Guppy 的集成 minimap2 aligner ,您可以立即節省 I / O 時間,并從 A100 用于 RNN 推斷的張量核心中獲益。通過在 DGX 上分別平衡八個 A100 GPU 和 256 CPU 線程的基址調用和對齊,這兩個進程可以完全重疊,以便與基址調用同時對齊讀取,不會對總運行時間造成影響(< 1 分鐘)。
這使 DGX-A100 上的 basecalling 和校準步驟的運行時間變為 2h 46m ,這也可以與測序本身重疊。這與 60 倍樣本的預期測序時間相似。
變異呼叫
變體調用是工作流的一部分,旨在識別新組裝個體基因組中與參考基因組不同的所有點。這包括掃描基因組的全部寬度,以尋找不同類型的變異。例如,這可能包括小的單堿基對變體,一直到覆蓋數千個堿基對的大結構變體。世界紀錄管道使用胡椒粉作為小變體,使用嗅探作為結構變體。
PEPPER Margin DeepVariant 方法旨在優化小變異,以實現納米孔測序產生的長讀。
- PEPPER 通過 RNN 識別候選變體, RNN 由兩個雙向、選通、循環單元層和一個線性轉換層組成。
- Margin 然后使用隱馬爾可夫模型方法進行一個稱為單倍型的過程,確定哪些變體是從母系或父系染色體一起遺傳的。它將此信息傳遞給 Google DeepVariant ,以最大限度地提高雜合子變體調用的準確性。
- DeepVariant 通過一個深度卷積神經網絡對最終變體進行分類,該網絡建立在 Inception v2 體系結構之上,專門適用于 DNA 讀取堆積輸入圖像。
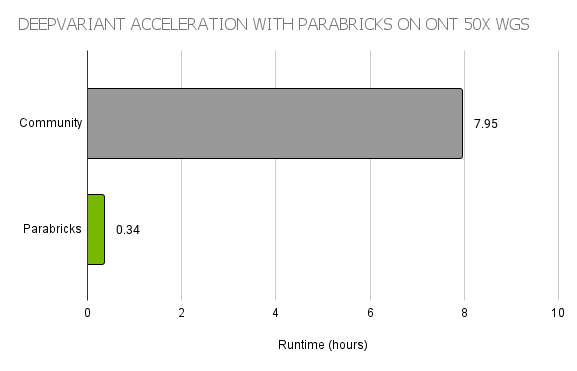
總的來說, PEPPER Margin DeepVariant 允許更快的 PEPPER 神經網絡掃描整個基因組尋找候選基因,然后使用更大的 DeepVariant 神經網絡對這些候選基因進行高精度的變異調用。為了加速這條管道,世界紀錄工作流使用了 Parabricks DeepVariant ,這是一種 GPU 加速的實現,比 CPU 上的開源版本快 20 倍以上(圖 2 )。
Clara 團隊通過修改 PEPPER Margin 以集成方式運行,按染色體分割數據,并在 GPU 上同時運行程序,進一步加快了速度。 PEPPER 還針對批量大小、工作人員數量和呼叫者數量等管道參數進行了優化,并對 PyTorch 進行了升級,以支持 NVIDIA 安培體系結構加速 RNN 推理瓶頸。
對于結構變量調用, Snifgles 升級為最近發布的 Snifgles 2 ,其效率要高得多,僅在 CPU 上的加速度為 38 倍。
所有這些改進使 DGX-A100 的多變量調用階段的運行時間達到 1h 24m 。
使用 NVIDIA DGX-A100 為實時測序供電
通過優化 DGX A100 的世界記錄 DNA 測序技術,NVIDIA Clara 團隊為實時測序提供了動力,簡化了單個服務器上的復雜工作流,并且在達到最先進性能的同時,將分析成本降低了 50% 以上。
這條管道將免費提供給研究人員。 立即請求訪問。
特色圖片由牛津納米孔技術公司提供