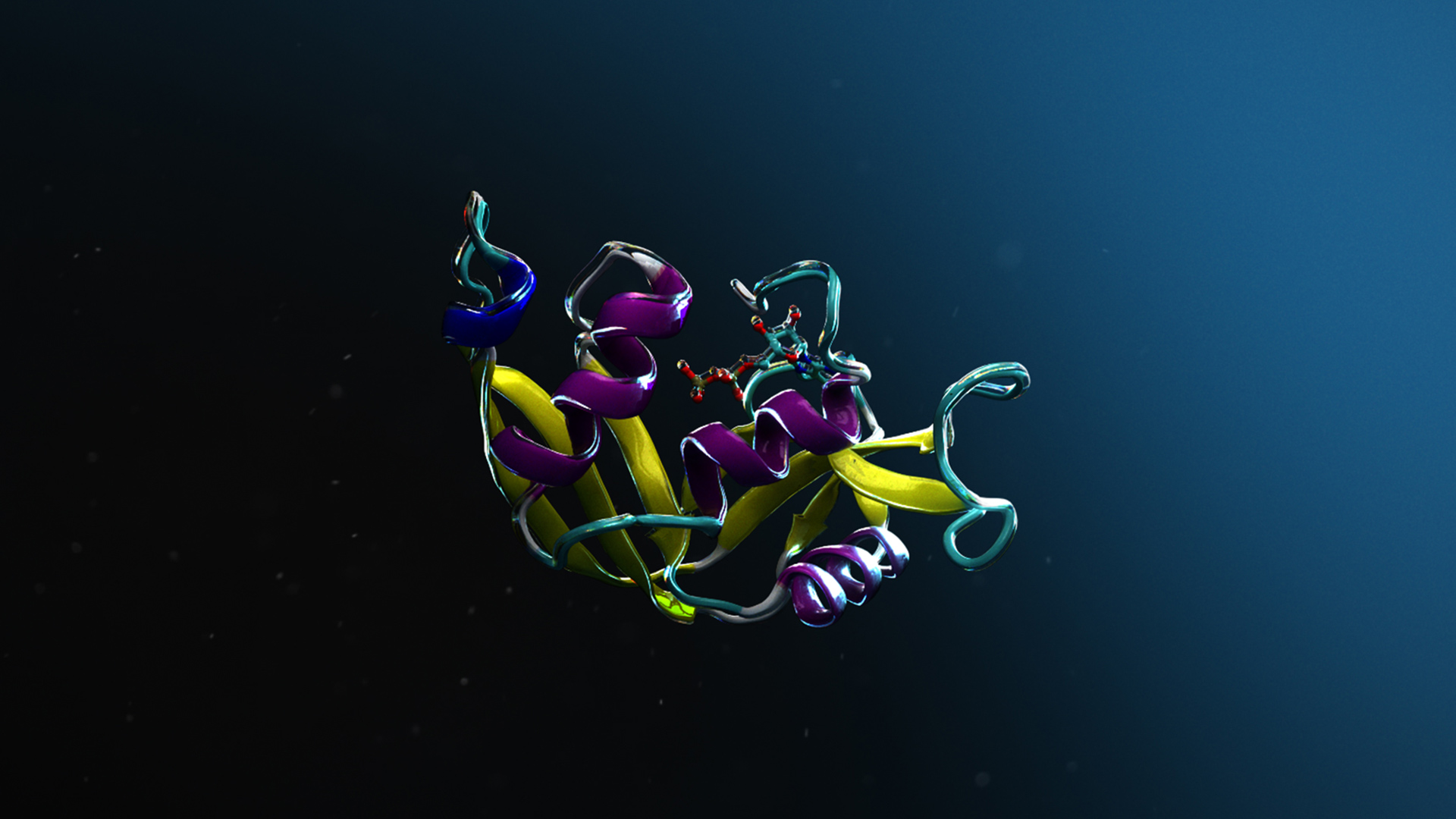
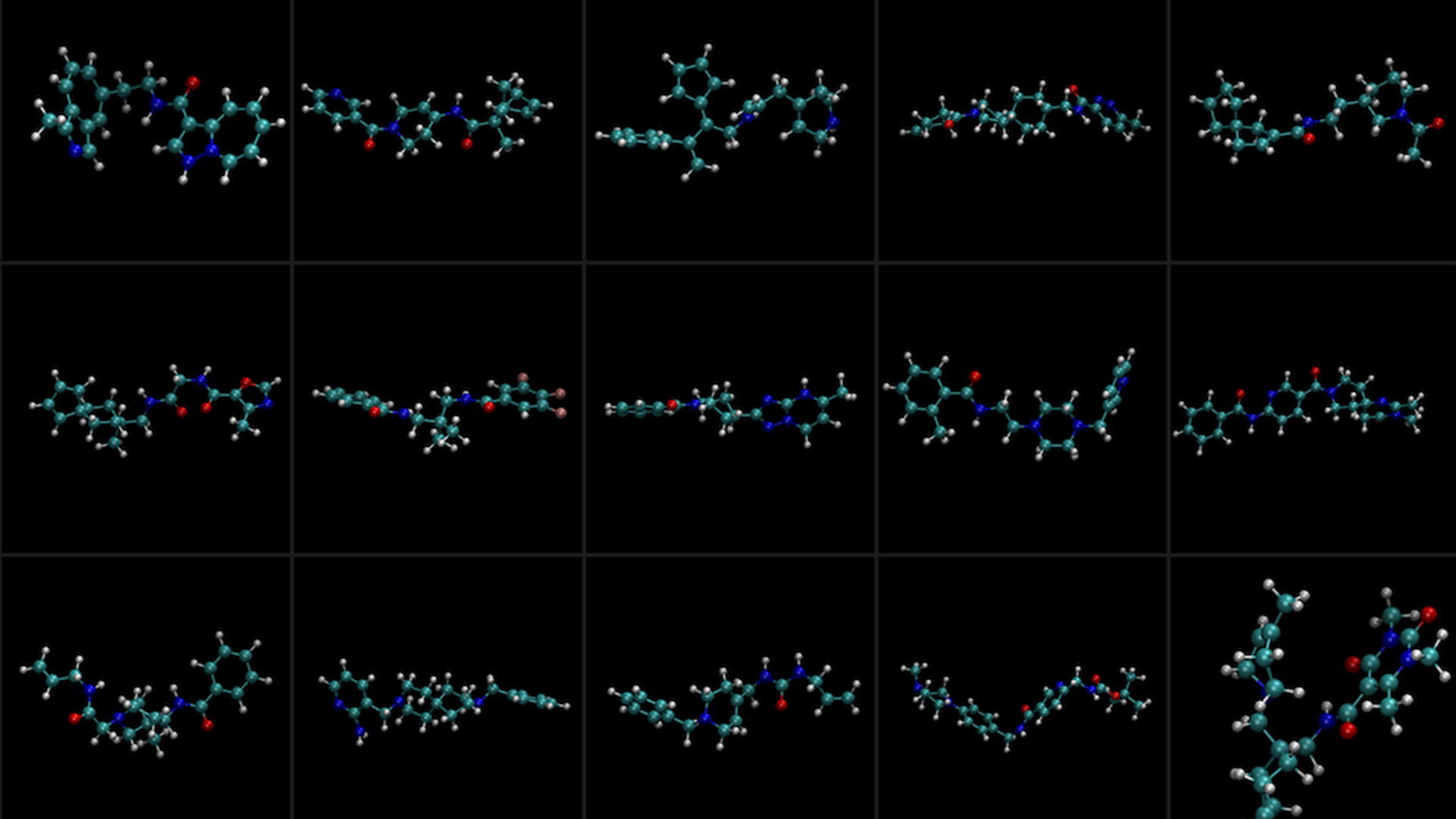
從細胞新陳代謝到工業制造,酶是眾多過程的重要生物催化劑。應用人工智能生成酶是一個令人興奮的研究領域,可直接應用于生命科學。在這些科學挑戰中取得進展對于進一步推動藥物研發、環境科學和生物工程的發展至關重要。
目前,地球上大量生命形式中只有一小部分進行了測序,這阻礙了機器學習算法在序列設計的復雜領域中的廣泛應用和泛化。改進的功能標記方法是酶研究的重要組成部分,能夠識別和表征新發現的酶的功能。這是了解復雜的生物過程和增強用于生成工作流程的數據的關鍵。
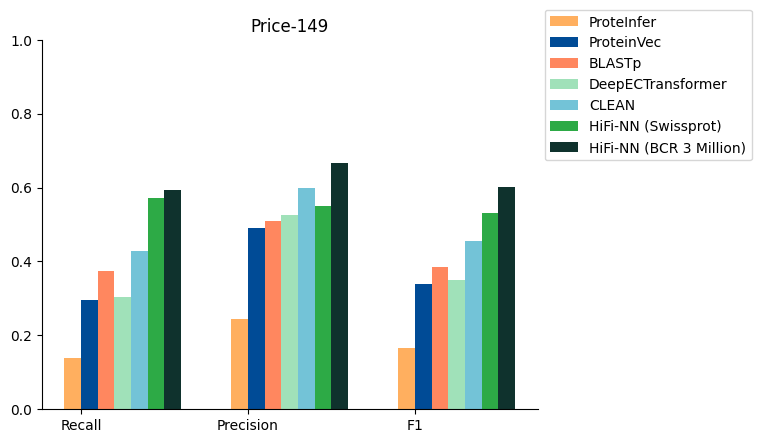
基地營研究,一家位于倫敦的 Bio-AI 公司,同時也是 NVIDIA 初創加速計劃 的成員,最近利用 NVIDIA GPU 訓練出了一種層次微調的最近鄰方法(HiFi-NN)。這種方法在召回率、精確度和 F1 分數方面相較現有模型有顯著提升,在酶標注任務上比先進的 SoTA 方法高出 15% 以上。
在全球探險活動中收集的獨特數據
Basecamp Research 可以應對整個生物技術行業中最復雜的生物設計挑戰。現有數據集存在重大缺陷:
- 代表性較小,僅涵蓋了地球上 0.0001% 的生命形式
- 沒有一致的元數據
- 在收集數據之前缺乏利益相關者的同意和參與
Basecamp 選擇通過與五大洲和 23 個國家 地區的自然公園建立生物多樣性合作伙伴關系來開發其專有的生物數據資源。他們派科學家進行全球探險,從最極端和非凡的生物群系中發現新的基因組、酶和生物關系。
在不到兩年的時間里,他們創建了 BaseGraph,這是自然生物多樣性的最大知識圖,包含超過 55 億個關系,每個蛋白質的基因組背景超過 70 千堿基。他們廣泛的長讀測序得到全面元數據收集的補充,使他們能夠將感興趣的蛋白質與特定反應和所需的工藝條件聯系起來。
Basecamp Research 的 AI 策略以數據為中心,原因有二:其專有數據可在快速商業化的算法環境中增強模型性能,并顯著彌補公開數據中缺乏多樣性的問題。
他們從頭開始構建的知識圖捕獲并重新創建了自然界中 40 億年蛋白質進化的復雜性。這一數據優勢使其 AI 和產品團隊能夠超越 SoTA 標注和設計模型,解決生物技術行業(從基因寫入療法到塑料降解)中的復雜設計挑戰。
在虛擬模式下,函數標注
為了應對蛋白質和酶的硅功能標注挑戰,Basecamp Research 的深度學習 (DL) 團隊開發了 HiFi-NN 搜索工具。HiFi-NN 利用酶委員會 (EC) 編號來標注蛋白質序列,而目前首選的生物信息學工具是 blastp,此外還包括其他 SoTA DL 模型,例如 CLEAN,它們在精度和召回方面的表現見表 1。
| 方法 | 召回 | 精度 | F1 分數 |
| ECPred | 0.0197 | 0.0197 | 0.0197 |
| DEEPpre | 0.0403 | 0.0415 | 0.0386 |
| DeepEC | 0.0724 | 0.1184 | 0.0846 |
| ProteInfer | 0.1382 | 0.2434 | 0.1662 |
| ProteinVec | 0.2961 | 0.4901 | 0.3378 |
| BLASTp | 0.3750 | 0.5083 | 0.3852 |
| DeepECtransformer | 0.3026 | 0.5263 | 0.3511 |
| CLEAN | 0.4671 | 0.5844 | 0.4947 |
| HiFi-NN (Swissprot) | 0.5724 | 0.5505 | 0.5304 |
| HiFi-NN (Swissprot+300 萬個精選序列) | 0.5921 | 0.6657 | 0.6015 |
BCR 3M 是指使用 Basecamp Research 的 BaseGraph 中 300 萬個不同環境的序列重新訓練的模型版本。
與顧問 Noelia Ferruz 和 Kevin Yang 合作開發的 HiFi-NN 在 2023 年 12 月的結構生物學機器學習研討會上獲得了著名 AI 會議 NeurIPS 的認可。
該模型使用 EC 編號的對比學習和用于自然增強的酶委員會標注系統的固有層次結構。該模型在 Lambda Labs 實例(CUDA 版本 11.8 和 NCCL 版本 2.14.3)上的 8 個 NVIDIA A100 GPU 上進行訓練,采用 PyTorch Lightning 進行分布式數據并行訓練。通過 Hydra 框架和 Weights and Biases 完成實驗管理和跟蹤。該模型擁有超過 300 萬個參數。
HiFi-NN 在 SoTA 標注方法中的出色性能歸功于使用 EC 編號系統中表示的酶功能的分層特性,以及使用 Basecamp 知識圖中的專有序列補充訓練集。
專有序列
作為對專有 Basecamp 序列的補充,HiFi-NN 來自五大洲的環境,oC 溫度范圍,以確保訓練集中盡可能多的序列和環境多樣性。因此,HiFi-NN 在基準測試數據集上的表現優于所有 SoTA 模型,并且在功能暗物質的蛋白質序列上的表現尤為出色,即那些與任何已知酶的相似性較低的序列。
實際上,Basecamp 團隊利用 HiFi-NN 對MGnify數據庫中之前未標注的微生物蛋白質進行了標注。
除了優于之前的所有標注模型外,HiFi-NN 還特別易于使用,并且能夠快速生成標注標簽。例如,它可以在單個 NVIDIA A100 GPU 上在 24 分鐘內標注整個人類蛋白質組。
Johnson Matthey 的快速酶識別
HiFi-NN 等突破增強了我們預測生物實體物理性質的能力,旨在減少使用資源密集型實驗室方法對候選對象進行廣泛篩選的需求。
Basecamp Research 與 FTSE100 化學公司 Johnson Matthey 合作,強調了計算進步在應對行業挑戰方面的重要性。在與該合作伙伴的一個項目中,研究人員在實驗室花費了一年半的時間測試數千種酶變體,但未能取得成功。
Johnson Matthey 的目標是找到能夠處理多個大型底物的具有廣泛特異性的酶,與處理較小的底物相比,這項任務更加復雜。在一周內,Basecamp Research 完全采用了硅技術來識別符合這些標準的酶(圖 2),從而將其定位為潛在的商業化。
該研究團隊的負責人對 Basecamp Research 快速發現和開發一種酶的能力表示贊賞,這是他們多年來一直無法完成的任務。這一成功為擴大各種酶開發計劃的協作努力奠定了基礎。
推動生命科學領域的 AI 發展
功能標注對研究人員發揮著關鍵作用,尤其是在以下實際場景中:
- 在藥物研發中,它通過闡明體內的酶相互作用來幫助創建有針對性的治療方法。
- 在工業生物技術領域,它支持針對特定工業應用定制的酶設計,從而推廣更環保的生產方法。
- 它還通過揭示不同物種中酶的發展軌跡,提供了有關進化生物學的重要見解。
從本質上講,由機器學習驅動的功能標記超越了單純的科學工具的作用。它是醫療健康和環境科學等各行各業創新和探索的催化劑。
Basecamp Research 與其他生命科學實體正在將其工作流程與 NVIDIA BioNeMo 集成, NVIDIA BioNeMo 是一個面向藥物研發的生成式 AI 平臺。該平臺簡化并加速了模型訓練。借助 BioNeMo,組織可以為各種目的定制和部署 AI 模型,包括 3D 蛋白質結構預測、從頭開始蛋白質和小分子生成、屬性預測和分子對接。
如果您對探索 BioNeMo 感興趣,可以通過 BioNeMo 框架的測試版或 API 搶先體驗計劃來獲得相關機會。欲了解更多信息,請訪問 NVIDIA BioNeMo 產品頁面。
?