即將發布的 NVIDIA Parabricks 是一套加速的基因組分析應用程序,它在加速測序比對和提高深度學習變體調用的準確性方面比以往任何時候都更進一步。該版本包括 PacBio 長讀數據的新工作流程,包括加速的 Minimap2 工具和谷歌的 DeepVariant ,用于對 PacBio 數據進行完整的 GPU 端到端分析。
NVIDIA Parabricks 可以免費使用,并提供付費企業支持選項。它包含各種優化的、基于人工智能的行業標準基因組工具,比基于 CPU 的工具提供高達 80 倍的加速,并將計算成本降低高達 50% 。與 CPU 上的約 24 小時相比,現在只需 16 分鐘即可分析 30 倍的全基因組,相當于每年在一臺服務器上分析多達 30000 個全基因組。
快速查看 Parabricks v4.1 的功能
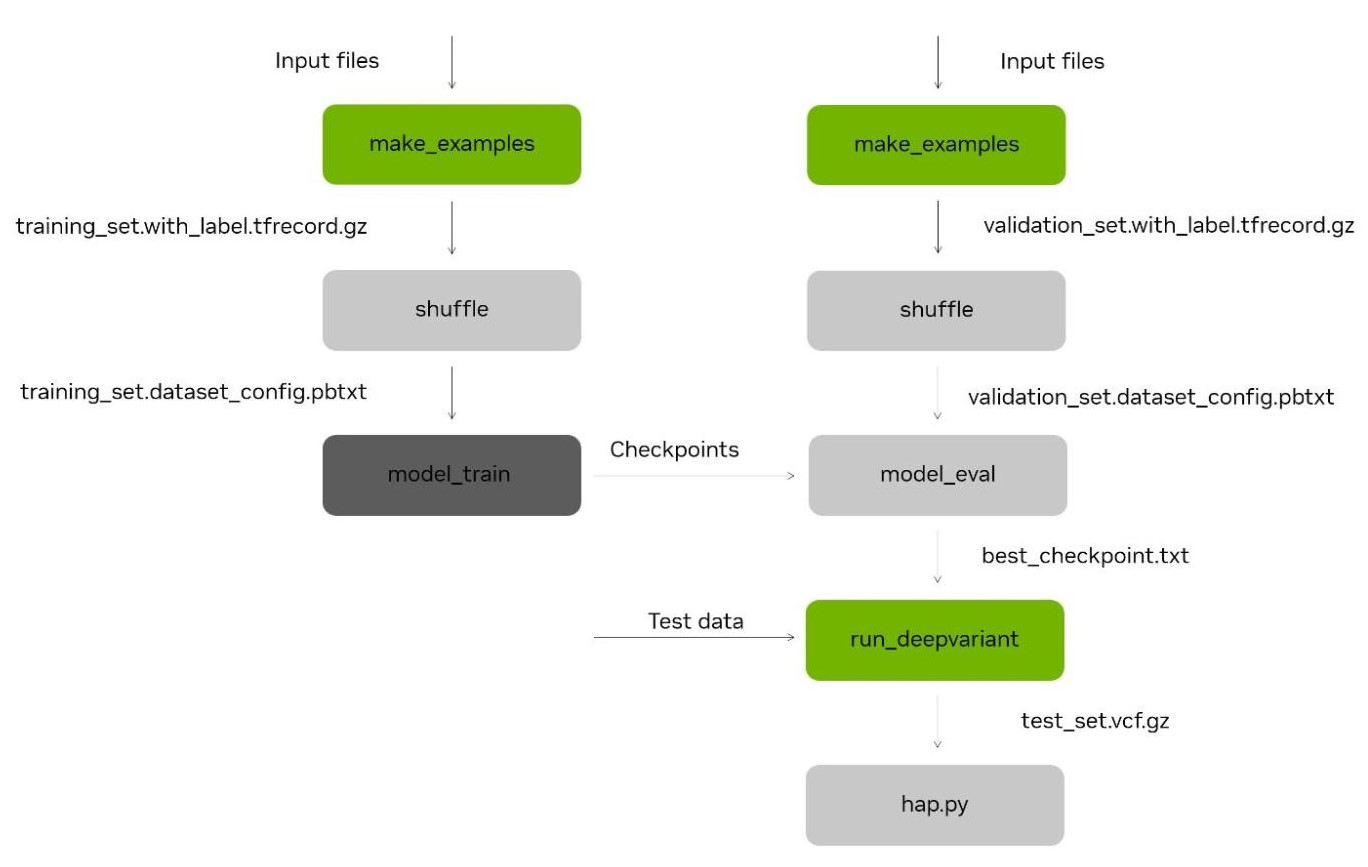
- 一種新的 DeepVariant 重新訓練工具,使任何人都能為自己的數據重新訓練或微調 DeepVariation ,從而實現更準確的變體調用(現已在 NGC 上提供)。
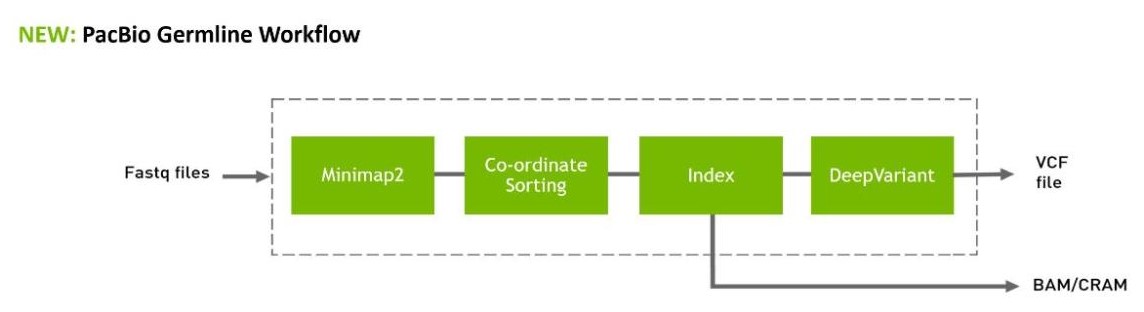
- PacBio 的端到端( FastQ 到 VCF )加速工作流,將在 GitHub 、 Terra.Bio 和其他云平臺上的 Parabricks 工作流中提供。
- 新的加速 Minimap2 工具,用于調整 PacBio 的長讀數。
- 用于 PacBio 數據的新加速 DeepVariant 變體調用程序,在 DGX 站[4xA100 GPU s]上運行 30 倍全基因組,運行時間為 8 分鐘。
- 與 v4.0 中的 21 分鐘和僅在 CPU – 上的約 24 小時相比, DGX A100 GPU [8xA100 GPU s]在 16 分鐘內進一步加速了 30 倍全基因組的短讀種系管道。
- 與新的 NVIDIA H100 GPU 兼容,其中包括強大的 DPX 指令,用于增強動態編程算法,如 Smith Waterman ,用于局部序列比對。
注冊以獲得 Parabricks 4.1 release 的通知,或嘗試 prerelease DeepVariant re-training tool 。
支持長讀分析
長讀測序,即對明顯較長的 DNA 片段進行測序的能力,與傳統的短讀測序相比具有多種固有優勢。最重要的是,這些讀數更容易被組裝到完整的基因組中。
較低水平的模糊性和比對誤差使長讀測序更好地用于基因組中更具挑戰性的部分(例如,高度重復的區域)或組裝基因組 de novo (沒有提供參考文獻)。
這為測序界帶來了許多改進,包括對結構變異(大插入、缺失、反轉、重復等)有了更多的了解。結構變異可能導致疾病,如盧·格里格病( ALS )、帕金森病和心臟病。
它還最終使科學界能夠端到端地完全完成人類參考基因組,即 2022 年發布的端粒到端粒( T2T )基因組。
PacBio 是長閱讀測序領域的杰出領導者。他們的技術產生長達 25 千堿基的讀取(相比之下,每次讀取的短讀取測序< 300 堿基)。他們還通過基于循環一致性測序的 HiFi 讀取技術和基于 transformer 的深度學習模型 DeepConsensus 的分析,突破了測序準確性的界限。
PacBio 的 Revio 長讀測序系統采用 NVIDIA GPU ,每年可將這種方法擴展到 1300 個人類全基因組。
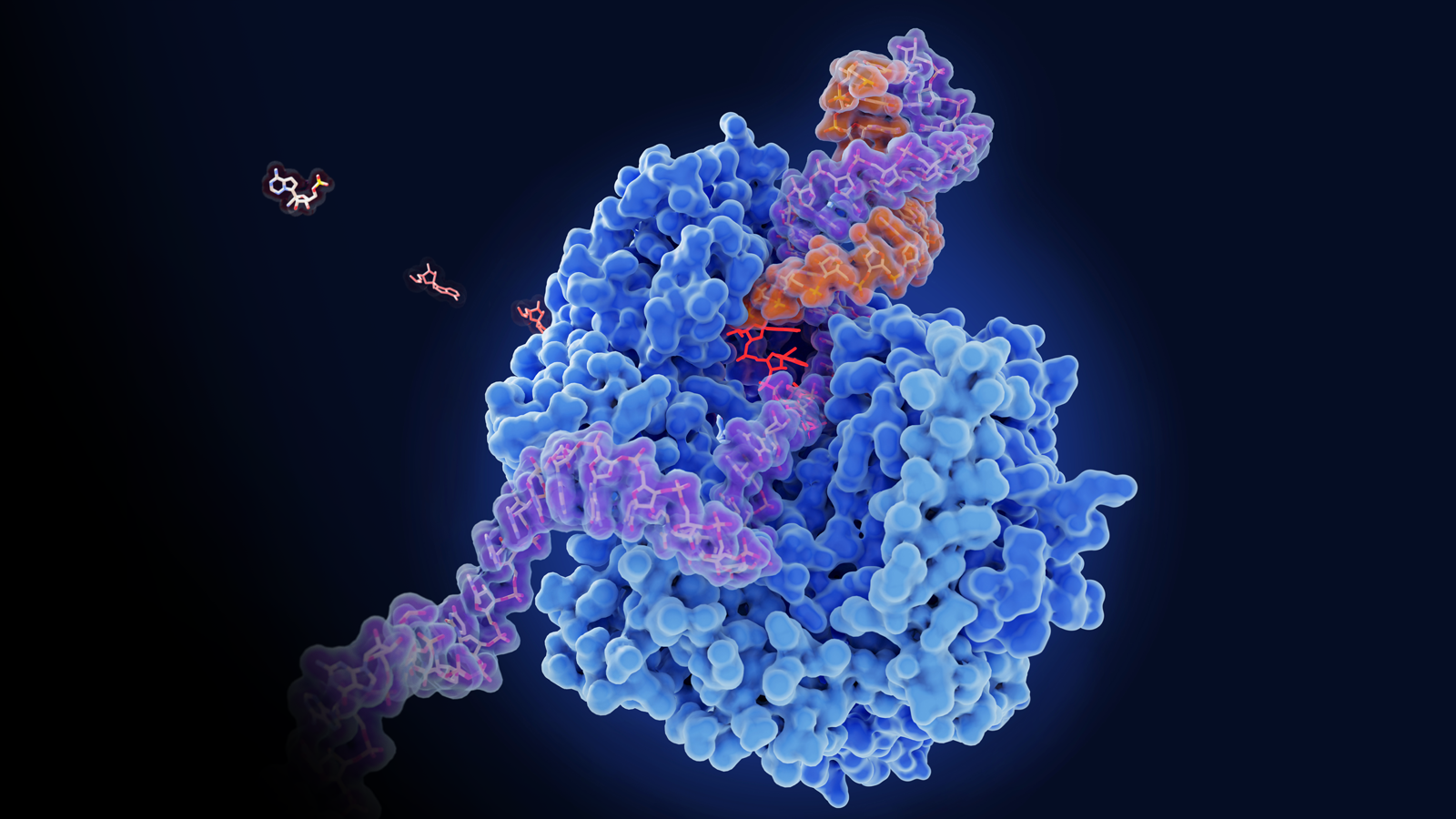
除此之外, NVIDIA Parabricks 4.1軟件可用于 GPU -與Minimap2的加速對齊,以及與DeepVariant的PacBio模型的變體調用,為PacBio數據提供完整的端到端工作流程。
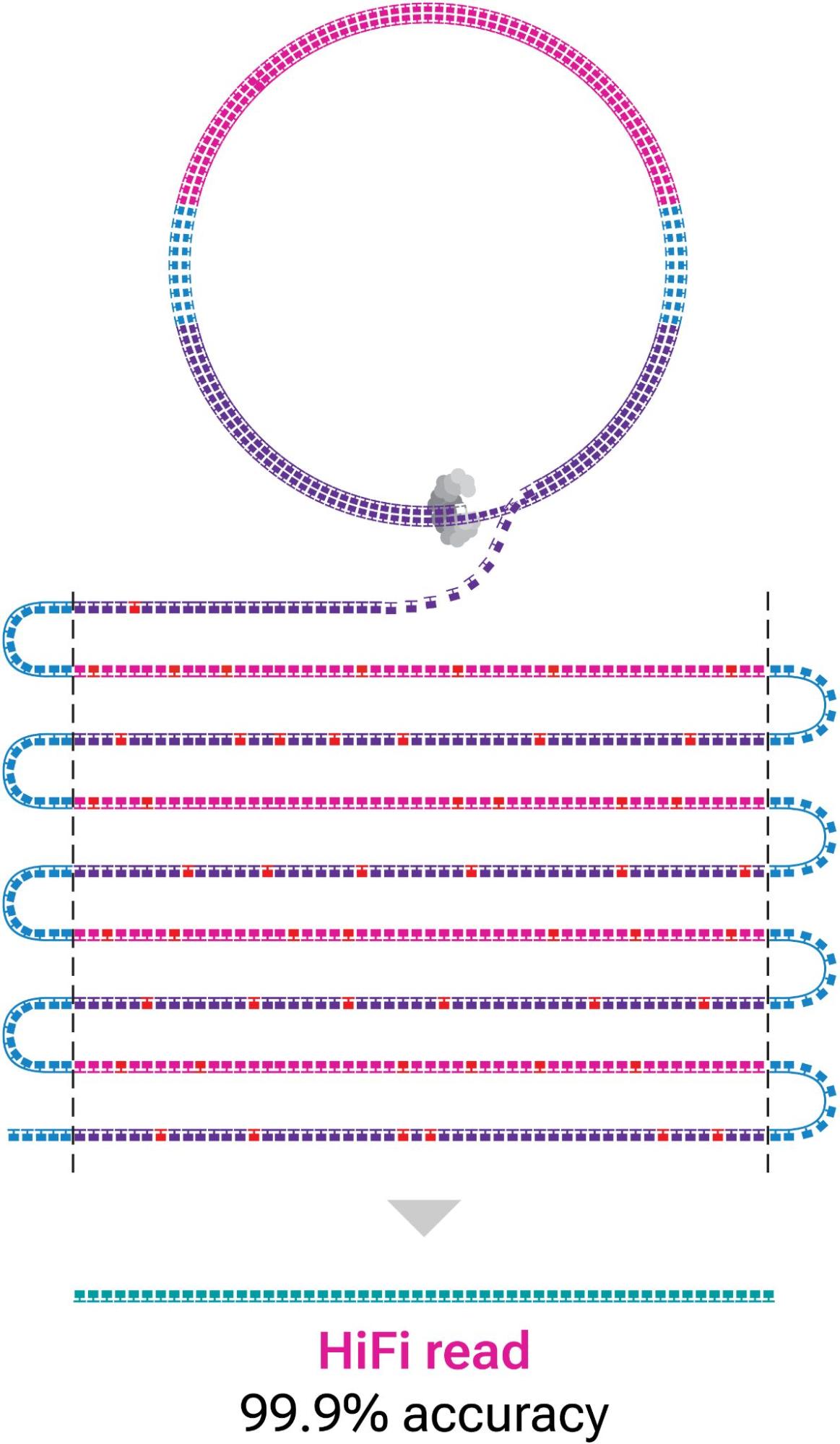
圖 3 顯示, HiFi 讀數是通過結合對 DNA 分子(子讀數)的多次連續觀測產生的,使單個 HiFi 讀取的準確性超過 99% 。
DeepVariant 使用 Parabricks 重新訓練
DeepVariant 是一個基于 CNN 的準確變體調用程序,用于短讀和長讀數據的種系工作流,作為 NVIDIA Parabricks 的一部分,在 GPU 上加速。 Parabricks 4.1 包括一個框架,用于重新訓練和微調基礎 CNN 模型,為分析工作流程帶來更準確的變體調用。
具體來說,這具有能夠將模型微調到單個數據集并識別后續數據中產生的任何非隨機偽影的優點。這已經成功地應用于測序儀級別,例如 Ultima 、 Singular 和 PacBio 都生產了自己的特定模型,并根據其獨特的誤差分布進行了訓練。
它也已應用于項目級別,例如 Regeneron Genetic Center’s exome sequencing as part of the UKBioBank project 。不同的實驗室通常使用不同版本的測序儀、濕實驗室試劑盒和試劑,并且通常有不同的實驗室流程。所有這些差異都可能在它們的樣本中引入微妙而獨特的人工制品。
通過使用 DeepVariant 基礎模型作為一個溫暖的開端,通過對少數瓶中基因組細胞系進行測序以進行訓練、測試和驗證,實施實驗室特定的微調可以是一個相對簡單的過程。
在 Regeneron 的情況下,使用單個 V100 GPU 訓練 12 小時,僅在一個樣本( HG001 )上訓練就足以看到模型收斂, 20% 的數據保留用于測試,第二個樣本( HG002 )用于驗證。這使得相對少量的數據在準確性上有了令人印象深刻的提高,例如將 INDEL 的孟德爾誤差率從 0.075 降低到 0.056 。
開始使用 NVIDIA Parabricks
NVIDIA Parabricks 在 NVIDIA NGC 和 DGX Cloud 以及其他云服務平臺和提供商上免費提供。今天通過 Parabricks collection on NGC 開始使用新的 DeepVariant 重新訓練框架。
請訪問 NVIDIA GTC 2023 加入我們,了解有關 training DeepVariant models with Parabricks 的更多信息。
要訪問本文中提到的其他功能,請注冊 Parabricks 4.1 release coming out in Q2 的通知。您可以在 Accelerated Genome Sequencing Analysis 頁面上了解更多關于 Parabricks 的信息,包括如何使用 guaranteed response times, priority security notifications, and access to AI experts from NVIDIA 通過 NVIDIA AI Enterprise 購買 enterprise support for Parabricks 。
加入我們的工程師 Parabricks 論壇,深入研究測序和深度變異調用。
?