許多組織正在使用 NVIDIA Clara Parabricks 對大型人群項目、重癥患者、臨床工作流程和癌癥基因組學項目進行快速人類基因組和外顯子分析。他們的工作旨在準確、快速地識別致病變種,與加速的下一代測序以及加速的基因組分析保持同步。
最近, 8 月和 9 月的兩份同行評審的科學出版物強調了 Clara Parabricks 在 de novo 和病原體工作流程中的速度、準確性和成本節約。
追蹤瘧疾傳播的基因組變異鑒定
普渡大學首席研究員喬瓦娜·卡皮博士和她的團隊試圖了解 Clara Parabricks 相對于瘧疾社區用于變種鑒定的現有方法的性能,以跟蹤瘧疾傳播并使用 1000 個瘧疾基因組監測抗瘧藥物耐藥性。
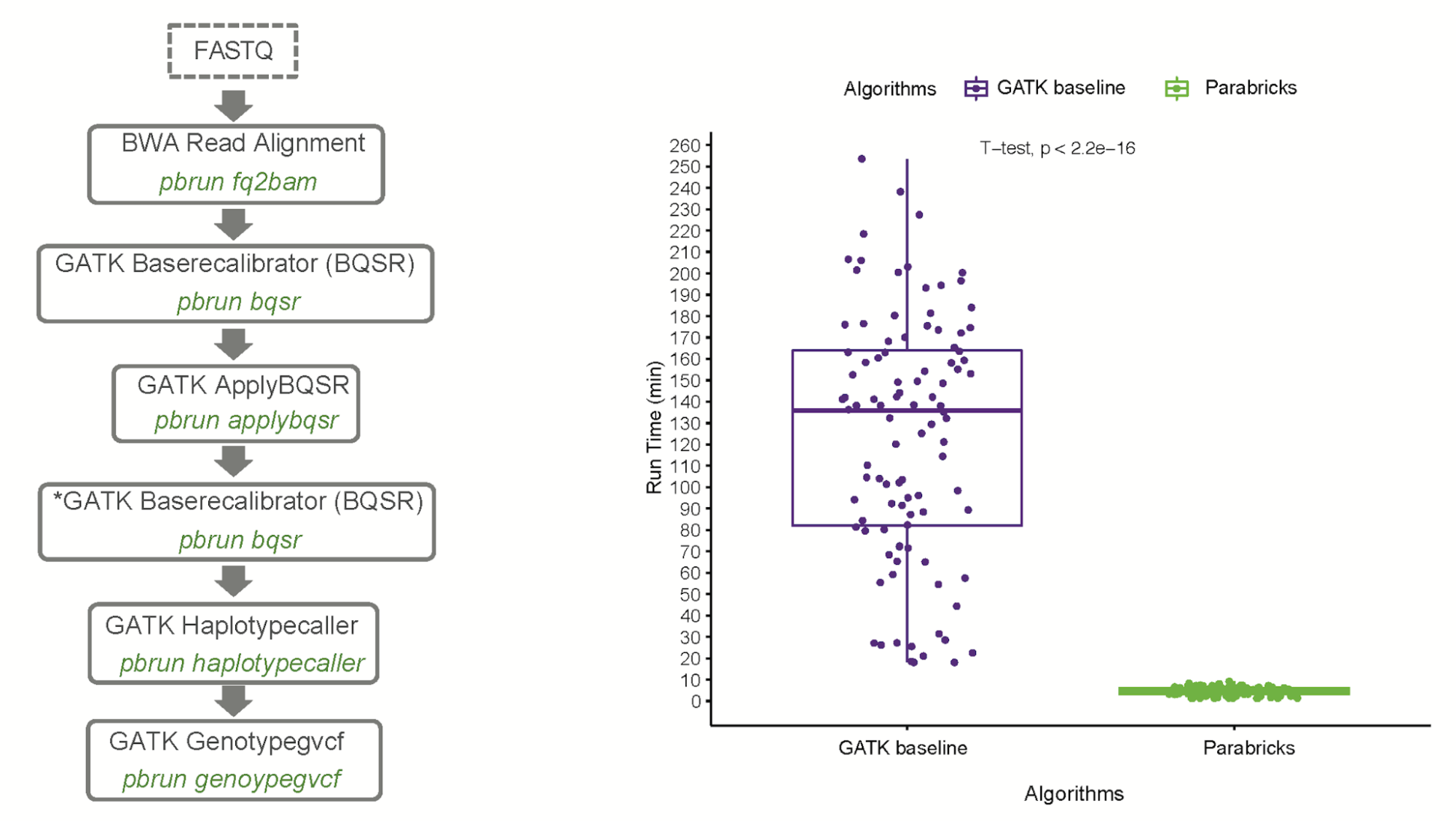
多年來一直在研究病原體基因組學的 Carpi 博士證明,與 CPU 傳統管道相比,分析速度提高了 27 倍,成本降低了 5 倍,同時準確率達到 99.9% 。瘧疾基因組相對較大( 24MB )且富含 AT ,這使得分析相當困難。 Carpi 博士使用了來自 MalariaGEN 財團的公開數據,這些數據是 Illumina 的原始數據。該研究發表在 A GPU-Accelerated Compute Framework for Pathogen Genomic Variant Identification to Aid Genomic Epidemiology of Infectious Disease: A Malaria Case Study 中,發表在 Briefings in Bioinformatics 中。
快速測序和分析全基因組病原體的能力有助于公共衛生官員了解疾病的傳播、耐藥性以及新變種的傳播性和嚴重性。世界衛生組織( WHO )報告 241 million cases of malaria in 2020 相比 2019 年的 2.27 億例, 2020 年估計有 627000 人死亡,比前一年增加 69000 人。
瘧疾是由 Plasmodium 寄生蟲引起的,這些寄生蟲通過受感染的雌性 Anopheles 蚊子叮咬傳播給人。非洲在全球瘧疾負擔中所占比例過高,五歲以下兒童占該地區總死亡人數的 80% 。
Carpi 博士指出,“能夠在不到五分鐘的時間內以 99.9% 以上的準確率以較低的成本為大規模全基因組 Plasmodium 研究生成分析就緒的變體輸出,顯著減少了大多數瘧疾基因組學項目目前面臨的計算瓶頸,并促進了流行國家的分散生物信息學分析。“訪問 GitHub 上的 malaria-parabricks-pipeline 下載此 Clara Parabricks 瘧疾工作流并了解更多信息。
在自閉癥患者中發現 de novo 變異
另外,來自圣路易斯華盛頓大學的 Tychele Turner 博士和她的團隊開發了一種快速基因組學工作流程,用于使用 GPU 加速的 Clara Parabricks 在自閉癥患者中發現 de novo 變異( DNVs )。特納博士是一位遺傳學家/基因組學家,對理解人類疾病的遺傳結構有著濃厚的興趣。她的實驗室專注于神經發育障礙的基因組學、基因組工作流程的優化以及應用新的基因組技術來理解疾病。這項研究發表在 De Novo Variant Calling Identifies Cancer Mutation Signatures in the 1000 Genomes Project 中,發表于 Human Mutation.
Turner 博士與 NVIDIA 基因組團隊密切合作,將她的三重分析整合到 NVIDIA Clara Parabricks 中。特納博士驚訝地看到,使用 NVIDIA Clara Parabricks 進行三重分析的周轉時間加快了 100 倍。在 GPU 上生成 DNV 的初始分析使用只有 4 個 GPU 的服務器需要 8.5 小時,而在 CPU 上需要 800 小時。當團隊在 GPU 上進一步并行化工作流程時,運行時間進一步縮短到不足一小時。
特納博士的大部分職業生涯都集中在 DNV 上,這是一種存在于兒童 DNA 中但不存在于父母 DNA 中的新變種。這些 DNV 可以通過對孩子和父母的 DNA 進行測序,然后進行比較分析(稱為三重分析)來評估。在普通人群中,每個個體約有 40 至 100 個 DNV ,大多數 DNV 不影響基因。
然而,當堿基對( a 、 T 、 C 、 G )中的單核苷酸變異( SNV )、小插入/缺失( indel )或結構變異( SV )改變基因并影響產生的蛋白質生產或功能時,通常會導致遺傳病。一些神經發育障礙就是這樣,在包括自閉癥、癲癇、智力殘疾和先天性心臟缺陷在內的表型中,患者體內蛋白質編碼 DNV 的富集已被確認。
這些快速的結果不僅為科學發現帶來了希望,也為特納博士對當天臨床結果的展望帶來了希望。為了確認基于 GPU 的新工作流程中 de novo 變體調用的準確性,該團隊利用 NVIDIA Clara Parabricks 研究了一個具有相同 DNA 的單卵孿生(也稱為同卵孿生)的家庭。
結果顯示,在基于 GPU 和以前基于 CPU 的工作流中, DNV 的數量相同,在這兩種情況下都發現了大約 20% 的 CpG 站點,這表明 NVIDIA Clara Parabricks 工作流產生了相同的結果,但速度快了 100 倍。這意味著他們的自閉癥基因組研究可以更快地完成,變異可以更快地被發現,并且有望更快地理解患者的見解。
特納博士表示,“ GPU 的利用使快速生物信息學分析能夠推進到一小時的基因組檢查。”
利用新的基于 GPU 的 DNV 基因組分析工作流程,該團隊繼續研究來自 1000 Genomes Project 的序列數據,這是一個國際研究聯合體,對來自非洲、東亞、南亞和歐洲人群的代表性隊列進行了測序。 1000 基因組項目旨在通過對來自世界各地 26 個群體的 2600 名個體進行測序,描述和表征人類基因組中發現的變異,作為研究遺傳多態性和表型之間關系的基礎。
最近,紐約基因組中心對這些個體進行了深度測序,并公開了數據。該人群包括 602 個沒有自閉癥的家庭。這是第一次有機會將沒有已知表型的 DNV 作為對照,以了解人群中 DNV 的水平,并將其與自閉癥隊列進行比較。
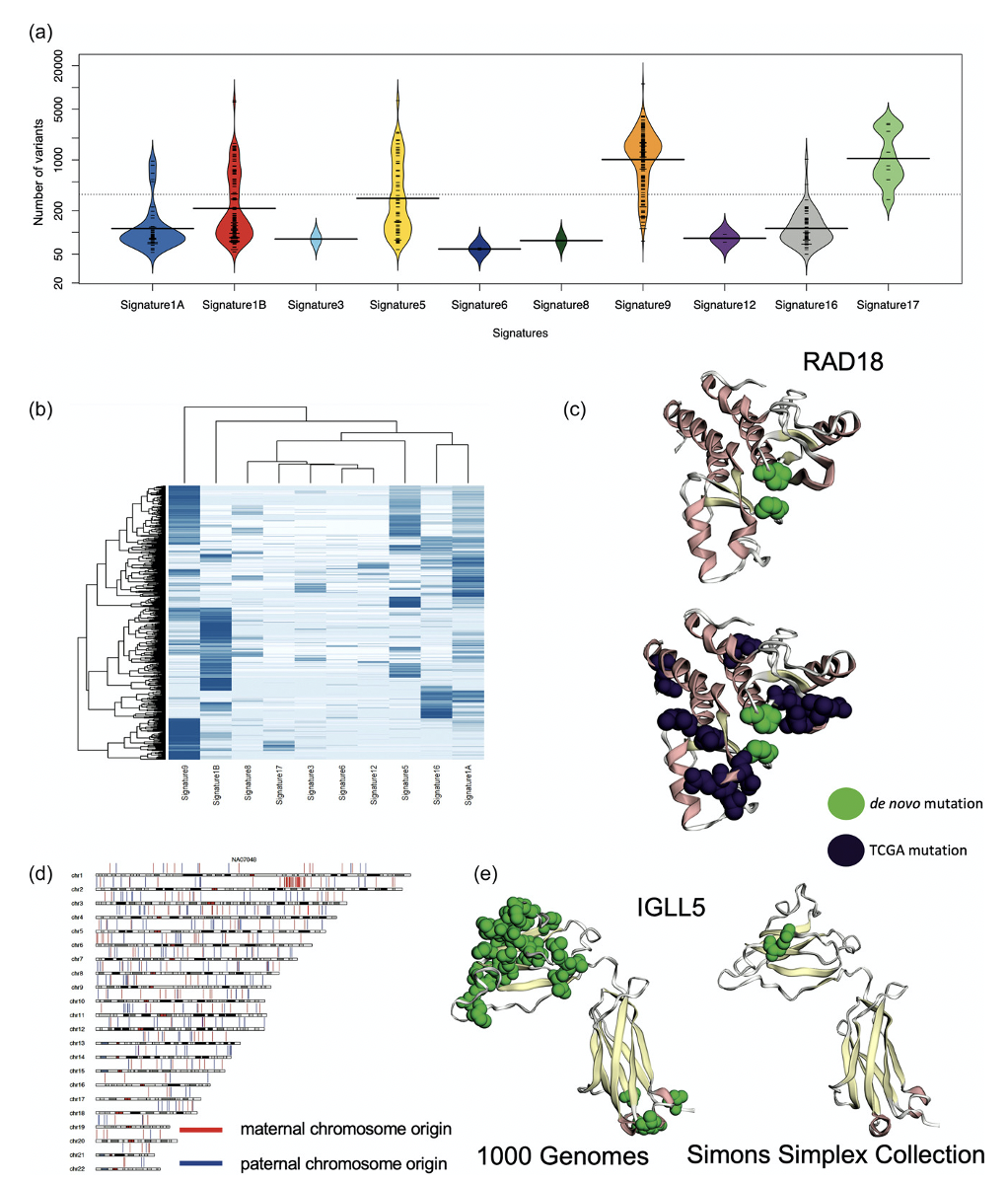
對 1000 名基因組計劃個體的 DNV 分析最終讓特納博士的團隊感到驚訝。他們發現 DNV 數量呈雙峰分布,峰值為 200 ,略大于預期,峰值為 2000 ,遠大于預期。特納博士查看了 1000 基因組計劃數據中的各個隊列,并注意到 CEU 人群是歐洲個體的隊列,研究時間更長,因此也培養了更多,可能導致更多的細胞系偽影。
在隊列中,有一名被確定為 NA12878 的個體進行了多次測序: 2012 年、 2013 年、 2018 年和 2020 年進行了兩次測序。特納博士表明, DNV 隨著時間的推移而增加。 2020 年的 DNV 最多,支持了 2020 年樣本中的細胞系偽影比 2012 年樣本更多的結論。該團隊得出結論,盡管 1000 基因組項目是基因組研究的一個極好的數據來源,但由于細胞系偽影的普遍存在,它可能不適合篩選患者對照的數據集。
盡管 1000 基因組計劃提供了關鍵的生物學和實踐見解,但只有 20% 的兒童擁有預期數量的 DNV ,大量證據表明過量的 DNV 是細胞系偽影。過量的 DNV 與 B 細胞淋巴瘤癌的突變特征相匹配,表明細胞系偽影不是以隨機方式累積的。
蛋白質編碼的 DNV 在 DNA 修復基因中被鑒定,并可能導致過量的 DNV 。 602 名個體的隊列對于已知在 B 細胞淋巴瘤中具有過量突變的 IGLL5 中的蛋白質編碼 DNV 具有重要意義,并且具有這些 DNV 的個體都具有大于 100 個 DNV 。蛋白質編碼 DNV 在臨床相關的變異位點中被識別,在使用該數據作為患者的二進制過濾集時需要謹慎。未來進行基因組測序的基因組研究應側重于基于家族的方法或利用直接從血液中提取的 DNA 建立良好的對照和參考數據庫。
特納博士評論道:“我的實驗室很高興開發出一種 de novo 變體調用工作流,該工作流利用 GPU ,使我們能夠快速分析近 4800 個全基因組測序的親子三人組,以獲得重要的生物學見解。”
為基因組研究提供動力的一套加速工具
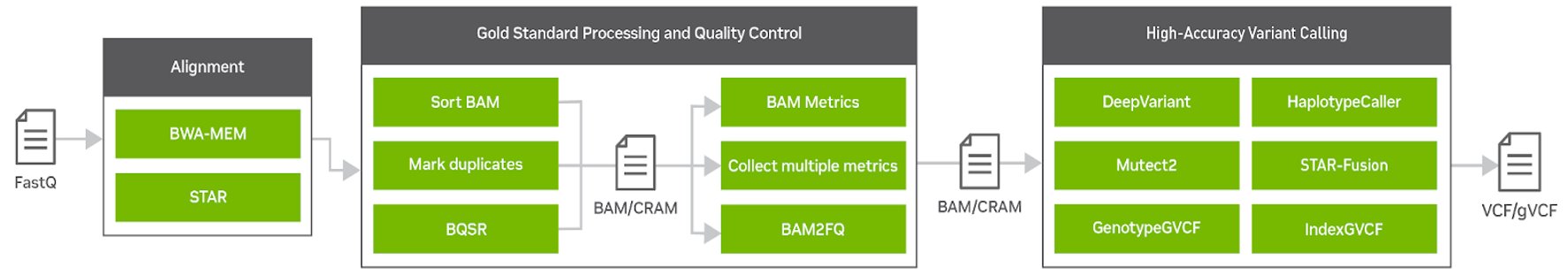
Clara Parabricks v4.0 是一個比以前版本更專注的基因組分析工具集,具有快速比對、金標準處理和高精度變體調用。它提供了自由無縫地交織 GPU 和 CPU 任務的靈活性,并優先考慮基因組學工作流程中最流行和瓶頸工具的 GPU 加速。 Clara Parabricks 還可以整合基因組學中的前沿深度學習方法。
您可以免費注冊 下載 Clara Parabricks 。您還可以請求一個免費的 Clara Parabricks NVIDIA LaunchPad Lab 演示,體驗用于外顯子組和全基因組數據集的種系和體細胞分析的加速行業標準工具。
有關 Clara Parabricks 的更多信息,包括可用工具的技術細節,請查看 Clara Parabricks 文檔?。
?