特別是借助 AlphaFold 和 RosettaFold 等突破性成果,生成式 AI 正在改變藥物研發以及生物科技公司和研究實驗室研究蛋白質結構的方式,從而在蛋白質相互作用方面獲得突破性見解。
蛋白質是動態實體。正如 AlphaFold2 及其在生物學和醫學領域的應用 所示,我們假設蛋白質的原生狀態僅通過其氨基酸序列而為人所知。然而,單個蛋白質可以根據環境采用多個不同的構象,例如溫度的變化。這些構象可以反映靈活的區域、蛋白質復合體內的相互作用,或活動和非活動狀態之間的過渡。
Protai 是 NVIDIA Inception 計劃的成員,旨在捕捉不同蛋白質狀態之間的結構變化,為特定作用機制 (MOA) 確定最精確的蛋白質結構,而不是滿足于可能與治療無關的一個構象。
Protai 正在利用質譜法 (Mass Spectrometry) 和人工智能 (AI) 開發精密醫學解決方案,為人類健康和社會帶來真正的改變,從而推動藥物研發的發展。Protai 平臺的核心是蛋白質結構預測管道,該管道集成了諾貝爾獎獲得的蛋白質結構算法、基于物理的工具和專有蛋白質組學數據。
為了在其平臺上加速 AI 推理,Protai 采用 NVIDIA NIM 微服務進行藥物研發 ,這是一套經過優化的生成式 AI 生物學模型。借助 NIM 微服務,Protai 在不影響準確性的情況下顯著提高了蛋白質結構預測的吞吐量和延遲。本文將深入探討 Protai 如何集成 NVIDIA NIM 微服務,為準確且可擴展的蛋白質結構預測提供支持,從而轉變他們的藥物研發方式。
蛋白質復雜結構預測背景
了解蛋白質復合體是結構生物學的基本支柱。蛋白質復合體是由兩條或更多關聯的多鏈組成的一組,它們相互作用并一起發揮作用,以執行特定的生物學活動。多聚蛋白質是多種相互作用蛋白質的組合,可推動關鍵的生物學過程,并且是藥物研發的關鍵目標。 雖然科學界正在通過實驗或計算技術在確定每種蛋白質單體的結構方面取得進展 ,但蛋白質復合體的數量呈指數級增長。這凸顯了預測算法的需求,以促進這些復合體的結構化工作。
AlphaFold-Multimer 通過實現多聚蛋白質結構的高質量計算預測來填補這一空白。這項創新以 AlphaFold 為基礎,利用深度學習來解讀蛋白質間的相互作用。我們將在下一節中提供有關 AlphaFold 算法、參數、輸出和部署的更多詳細信息。
Protai 采用了一種多面的方法。其中一個關鍵策略是增強結構預測模型 (例如 AlphaFold-Multimer) 的采樣,以考慮結構偏移。此外,Protai 還能生成獨特的 交聯質譜 (XL-MS) 數據,以識別能夠揭示不同狀態下特定蛋白質結構的鏈接體。
XL-MS 是一種功能強大的實驗技術,可使用化學交叉鏈接劑以共價方式將蛋白質內或蛋白質之間的特定氨基酸殘基結合,從而捕獲空間接近性和交互位點。這些交叉連接的區域提供了寶貴的距離限制,從而能夠更精確地映射蛋白質構象和相互作用。通過將這些通過實驗衍生的限制與先進的采樣技術和分子動力學模擬相結合,研究人員可以生成超出目前公共領域可用的蛋白質結構。

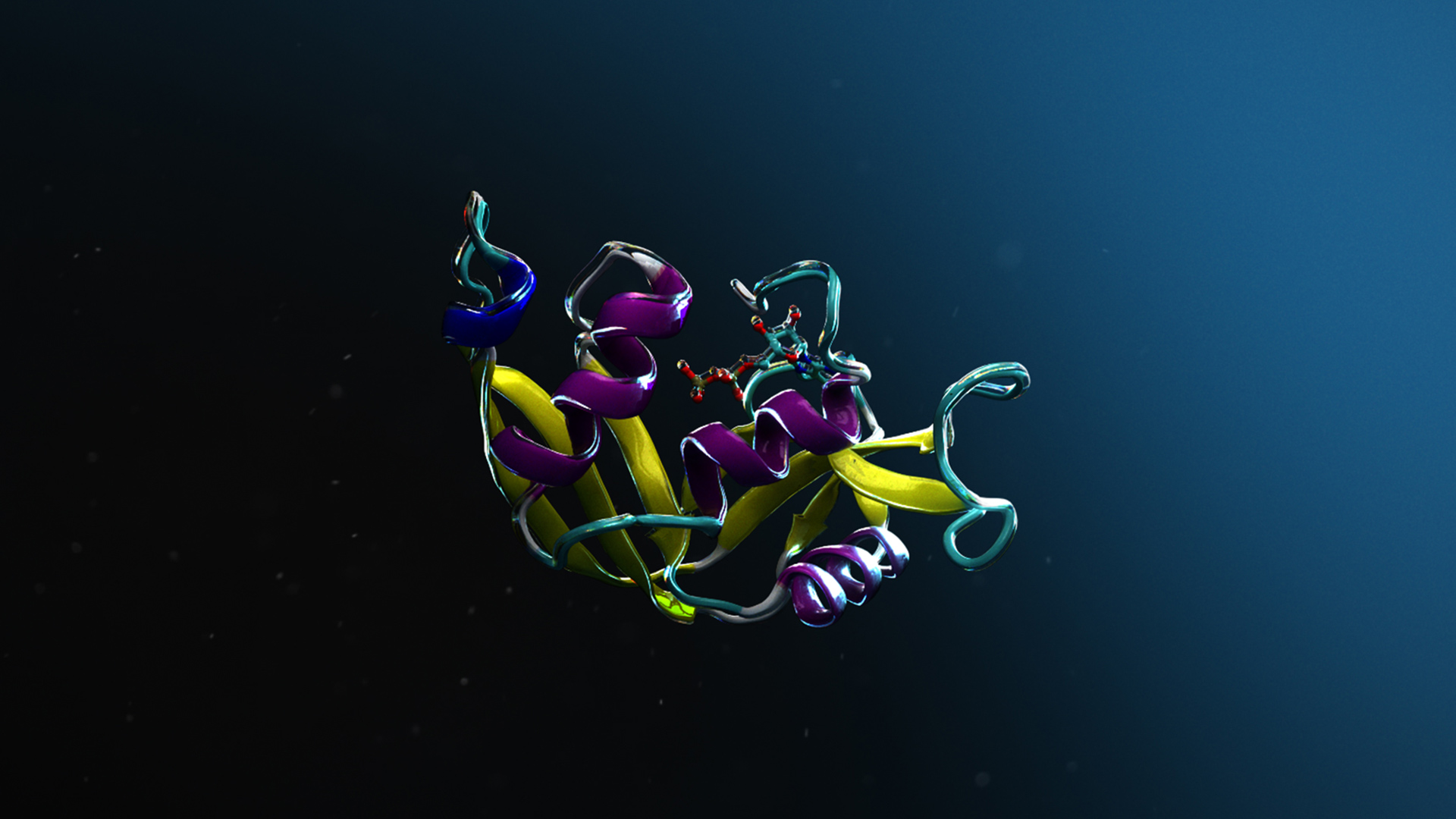
案例研究:預測 H3-H4 蛋白質復合體
為說明 Protai 的功能,本節研究了涉及組蛋白 H3 和 H4 的蛋白質復合體的預測。這種復合體在確定 DNA 能否獲得轉錄因子和 RNA 聚合酶方面發揮著至關重要的作用,同時還有助于在修復過程中提高 DNA 穩定性。
使用 AlphaFold2-Multimer NIM ,Protai 生成了 H3-H4 復合體的結構預測。生成的結構按置信度進行顏色編碼,以直觀方式表示預測準確性。對于包含在 AlphaFold 訓練集中的蛋白質,預測往往具有高置信度,但靈活性和獨特特征可能會因特定構象而異。

為了完善這些預測,Protai 利用 XL-MS 數據,識別了 H3 和 H4 之間的三種蛋白質間鏈接體。這些連接器可充當實驗錨定器,使 Protai 能夠驗證預測結構或揭示新的潛在蛋白質狀態。在本例中,前五個排名預測中,三種連接器得到了一致的保持。在這兩種蛋白質的高置信度和低置信度殘基區域之間確定了一個額外的連接器,這凸顯了進一步優化和改進預測的機會。

借助 NVIDIA NIM 進行 AlphaFold-Multimer 部署
為了支持 AlphaFold-Multimer 和其他基于 LLM 的工具的可擴展和優化部署,Protai 利用了 NVIDIA NIM。NIM 提供預先優化的推理容器,可在 NVIDIA GPU 上無縫部署。無論是在本地還是云端運行,這些容器均可確保提供先進的性能。AlphaFold 本身無法利用多個 GPU 進行單個預測。但是,NIM 微服務支持并行運行多個推理任務,從而顯著減少預測多個蛋白質復合體所需的總體時間。(對于大型復合體,預測可能需要 24 小時以上。)
該工作流由以下關鍵階段組成:
- 多序列對齊 (MSA):AnMSA 可識別保守區域以及相互作用的蛋白質之間的共同進化信號,為預測提供基礎。傳統的 MSA 工具依賴于基于 CPU 的實現,這雖然在順序處理方面有效,但無法與 GPU 并行處理能力相匹配。?AlphaFold2 NIM?使用?MMseqs2?,這是一款 GPU 優化的序列搜索和聚類套件,能夠以驚人的速度實現高效比較。
- 蛋白質相互作用建模 :AlphaFold-Multimer 使用基于 AlphaFold2 Transformer 架構的修改版,針對鏈間交互、增強的配對表示、跨鏈建模和多聚體特定損失函數進行微調,以預測蛋白質復雜結構。
- 結構細化: 預測結構經過細化,以確保準確性和物理合理性,同時納入立體化學限制和實驗基準。

AlphaFold2-Multimer NIM 提供多個端點,可將推理過程分為 CPU 密集型任務和 GPU 密集型任務,從而降低計算成本:
- protein-structure/alphafold2/multimer/ predict-structure-from-sequences:從序列 (端到端) 進行完整結構預測
- protein-structure/alphafold2/multimer/predict-MSA-from-sequences:從序列進行 MSA 計算 (CPU 密集型)
- protein-structure/alphafold2/multimer/predict-structure-from-MSA:通過預先計算的 MSA 進行結構預測(GPU-intensive)
硬件的選擇取決于蛋白質復合體的大小
- 短序列:32 GB 的 GPU 就足夠了。
- 更大的配合物 (> 3000 個殘基):需要 NVIDIA H100 或 A100 GPUs 來實現出色性能。
在上述用例中,Protai 在 NVIDIA L4 GPU 上自行部署了 AlphaFold2-Multimer NIM,因為這兩種蛋白質很小。
參數?
AlphaFold2-Multimer NIM 具有以下參數:
- 序列: 定義將用于執行多聚結構預測的目標蛋白質鏈。
- algorithm: 指定用于搜索同源序列的算法 。 jackhmmer 是一種廣泛使用的 MSA 生成算法 ,可識別進化相關序列,幫助模型學習蛋白質間的相互作用。
- e_value:用于識別數據庫中同源序列的閾值。較低的值表示更嚴格的匹配標準。
- 迭代:定義 MSA 算法執行的 迭代次數?(限制運行時間)。
- databases : 指定查詢準確的 MSA 所需的進化信息的 數據庫?。
- relax_prediction:表示是否應細化預測結構。通過優化鍵長、角度和立體化學約束,確保最終結構的物理合理性。
輸出?
蛋白質數據庫(PDB)格式的 AlphaFold2-Multimer NIM 輸出包含有關預測蛋白質多聚體的原子級結構信息。在輸出文件中,蛋白質的每個原子都使用符合 PDB 格式規范的結構化格式進行描述。

使用 AlphaFold2-Multimer NIM 的給定 PDB 行說明如下:
- 鏈標識符: 用于識別輸出中的蛋白質鏈,例如,由兩種蛋白質組成的蛋白質復合體至少具有兩條鏈。
- 坐標 (X,Y,Z): 原子在 ? 中的 3D 空間坐標。
- 占用 :在此位置觀察到該原子的概率。范圍從 0.00 (完全不存在) 到 1.00 (始終存在) 不等。在實驗數據中,如果原子在晶體結構中以多個構象出現,則其 占用 率可能小于 1.00 (例如。帶有多個旋轉器 (替代構象) 的側鏈在所有構象中的 占用 率求和為 1.00)。在 AlphaFold 預測中,所有原子的 占用 率都設置為 1,因為它為蛋白質提供了一個最有可能的結構。
- B 系數: 衡量結構中原子位移或靈活性的指標。值越高表示動作越多。在實驗數據中,它可以表示熱運動或障礙。在 AlphaFold 輸出中,它被從預測局部距離差分測試 (pLDDT) 中獲得的置信度分數所取代。pLDDT 是由 AlphaFold 提供的每殘差置信度分數,范圍從 0 到 100。
結束語?
Protai 的結構預測工作流將 AlphaFold2-Multimer NIM 與通過實驗識別的獨特 XL-MS 連接器相結合。通過利用 NVIDIA 優化的 AI 基礎架構,Protai 加速了預測并提高了可擴展性。這有助于探索之前無法接觸到的蛋白質相互作用,為藥物研發和精準醫療開辟了新的前沿。
H3-H4 測試用例展示了 Protai 的整合方法 (合并結構預測和 XL-MS 數據) 如何深入了解蛋白質構象和動力學,這對于理解生物功能和治療應用至關重要。
隨著生成式 AI 的不斷發展,NVIDIA NIM 和 NVIDIA BioNeMo Framework 等工具將在發揮計算生物學的全部潛力方面發揮關鍵作用。Protai 仍然致力于整合先進的 AI 技術,使我們離未來更近一步,在未來,救命藥物的開發速度將比以往更快、更高效、更精確。
您可以試用用于 藥物研發的全套 NIM 微服務 ,包括用于生成式蛋白質粘結劑設計的 NVIDIA BioNeMo Blueprint 和用于生成式虛擬篩選的 NVIDIA BioNeMo Blueprint。此外,您還可以使用 開源 BioNeMo Framework 訓練自己的生物學模型。最后,學習如何使用 GPU 加速的 MMseqs2 提升 AlphaFold2 蛋白質結構預測 。
?