AI 已從實驗好奇心發展為生物學研究的驅動力。 深度學習算法、海量組學數據集和自動化實驗室工作流程的融合使 科學家能夠解決一度被認為棘手的問題 (從快速蛋白質結構預測到生成式藥物設計),從而增加了科學家對 AI 素養的需求。在這一勢頭下,我們正處于下一次范式轉變的邊緣:專為生物學打造的強大 AI 基礎模型的出現。
這些新模型有望將不同的數據源 (基因組序列、RNA 和蛋白質組譜,在某些情況下還包括科學文獻) 統一為分子、細胞和系統級別上對生命的統一、一致的理解。學習生物學的語言和結構為變革性應用打開了大門,例如更智能的藥物發現、合理的酶設計和疾病機制闡明。
在我們為下一波 AI 驅動的突破做好準備之際,這些基礎模型顯然不僅能夠加速進展,還將重新定義生物學研究的可能性。
- 使用/NVIDIA/bionemo-examples 示例 notebook 免費將 Evo 2 測試為 NVIDIA BioNeMo NIM 微服務 。
- 探索蛋白質設計的完整參考工作流程 。
- 立即開始在 BioNeMo 框架 中使用您的數據訓練 Evo 2
- 及時了解 NVIDIA BioNeMo 平臺的最新動態。
序列建模和設計從分子規模到基因組規模的飛躍
2024 年 11 月推出的 首個 Evo 模型 是基因組研究領域的一個突破性里程碑,它引入了能夠分析和生成跨 DNA、RNA 和蛋白質的生物序列的基礎模型。
在發布 Evo 時,大多數模型都被限制在單一模式或簡短背景下運行,而眾所周知,它能夠使用統一的方法跨規模 (從分子到基因組) 操作。Evo 基于 270 萬個原核細胞和噬菌體基因組 (包含 300 億個核酸令牌) 進行訓練,在許多生物進化和功能任務中提供了單核酸分辨率。
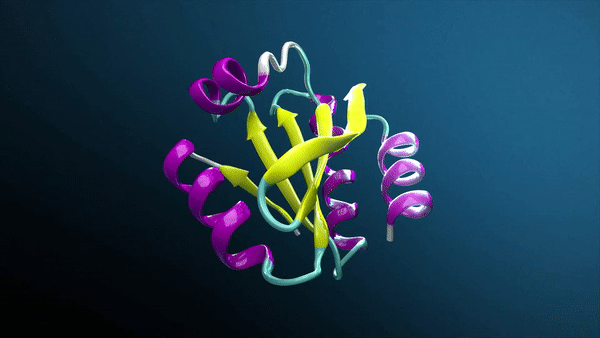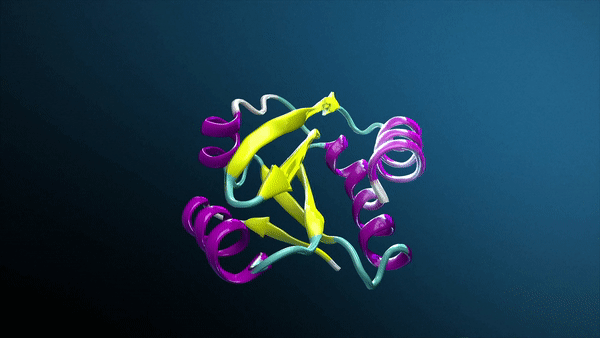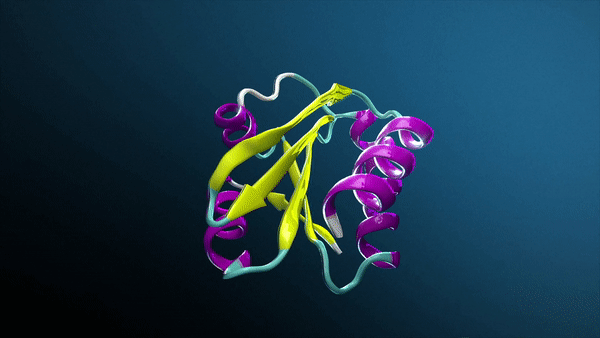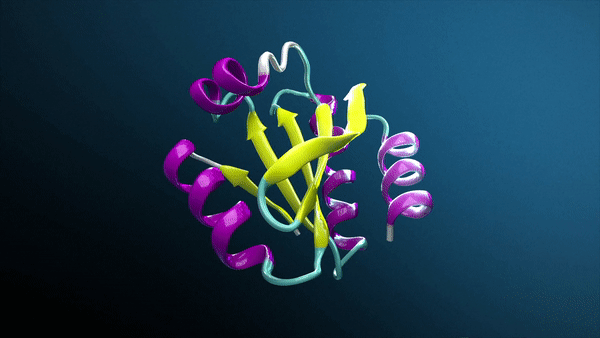
Evo 成功的核心是其創新的 StripedHyena 架構 (圖 1),這是一個結合了 29 個 Hyena 層的混合模型,這是一種新型深度學習架構,旨在處理長序列信息,而無需依賴 Transformer 架構中常見的傳統注意機制。相反,它使用卷積過濾器和門的組合。
這種設計克服了傳統 Transformer 模型的限制,使 Evo 能夠高效處理多達 131,072 個令牌的長上下文。最終,該模型能夠將微小的序列變化與系統級和有機體級的影響聯系起來,彌合分子生物學與進化基因組學之間的差距。

Evo 的預測功能為生物建模樹立了新的標準。它在多項零樣本任務中取得了具有競爭力的表現,包括預測突變對蛋白質、非編碼 RNAs 和調控 DNA 的適應性影響,為合成生物學和精準醫學提供寶貴見解。
Evo 還展示了非凡的生成功能,設計了功能齊全的 CRISPR-Cas 系統和轉座子。這些輸出經過實驗驗證,證明了 Evo 可以預測和設計具有真實效用的新型生物系統。
Evo 代表著將多模態和多尺度生物理解集成到單個模型中的顯著進步 。它能夠生成基因組級序列并預測整個基因組的基因本質,這標志著我們分析和工程生命的能力實現了飛躍。
Evo 的里程碑式發展不僅體現在技術成就上,還體現在愿景上。這一統一框架將生物學的龐大復雜性與尖端 AI 相結合,加速了生命科學領域的發現和創新。
學習進化過程中的生命語言
Evo 2 是基因組建模領域這一系列研究的新一代產品,基于 Evo 在擴展數據、增強架構和卓越性能方面取得的成功而構建。
Evo 2 可以深入了解三種基本生物分子 (DNA、RNA 和蛋白質) 以及生命的所有三個領域:真核生物 (Eukarya)、原核生物 (Prokarya) 和古菌 (Archaea)。此訓練數據集基于來自 15,032 個真核基因組和 113,379 個原核基因組的 8.85T 核酸數據集進行訓練,涵蓋不同物種,與僅關注原核基因組的 Evo 相比,實現了前所未有的跨物種泛化,并顯著拓寬了其范圍。
Evo 2 使用經過改進的全新 StripedHyena 2 架構,該架構可將參數擴展至 40B 個,從而提高模型的訓練效率,以及使用 1M 個令牌的上下文長度捕獲遠程依賴項的能力。 StripedHyena 2 采用基于卷積的多混合設計,其訓練速度明顯快于 Transformers 和其他使用線性注意力或狀態空間模型的混合模型。
最大的 Evo 2 模型使用 AWS 上的 NVIDIA DGX Cloud 使用 2,048 個 NVIDIA H100 GPUs 進行訓練。作為 NVIDIA 與 Arc 合作 的一部分,他們獲得了對這個高性能、完全托管的 AI 平臺的訪問權限,該平臺利用 NVIDIA AI 軟件和專業知識針對大規模分布式訓練進行了優化。
這些進步標志著 Evo 的 7B 參數和 131,000 個令牌的上下文長度有了顯著增加,使 Evo 2 成為多模態和多尺度生物建模領域的領導者 (表 1)。
| 特征 | Evo | Evo 2 |
| 基因組訓練數據 | 細菌 + bacteriophage(300B 核酸) | 所有生命領域 + bacteriophage(9T nucleotides) |
| 模型參數 | 70 億 | 70 億 400 億 |
| 上下文長度 | 131072 個令牌 | 最多 1048576 個令牌 |
| 模式 | DNA、RNA、蛋白質 | DNA、RNA、蛋白質 |
| 安全性 | 不包括真核生物病毒 | 不包括真核生物病毒 |
| 應用 | 跨物種任務受限 | 廣泛的跨物種應用 |
Evo 2 的擴展訓練數據和優化架構使其能夠在各種生物應用中表現卓越。其多模態設計集成了 DNA、RNA 和蛋白質數據,可在執行突變影響預測和基因組標注等任務時實現零采樣性能。Evo 2 還通過納入真核基因組從根本上改進了 Evo,使人們能夠更深入地了解人類疾病、農業和環境科學。
Evo 2 的預測能力優于各種任務的專業模型:
- 變體影響分析 :在零樣本預測物種突變 (包括人類和非編碼變體) 的功能性影響方面實現出色的準確性。
- 基因本質 :識別原核和真核基因組中的基本基因,并通過實驗數據集進行驗證,彌合分子和系統生物學任務之間的差距。
- 生成功能 :設計復雜的生物系統(例如基因組級的原核細胞序列和真核細胞序列),以及染色質可訪問性的可控設計,展示具有現實世界適用性的生物設計新功能。
使用 NVIDIA Evo 2 NIM 微服務
NVIDIA Evo 2 NIM 微服務 可用于生成各種生物序列,其 API 可提供用于調整標記化、采樣和溫度參數的設置:
# Define JSON example human L1 retrotransposable element sequenceexample = { # nucleotide sequence to be analyzed "sequence": "GAATAGGAACAGCTCCGGTCTACAGCTCCCAGCGTGAGCGACGCAGAAGACGGTGATTTCTGCATTTCCATCTGAGGTACCGGGTTCATCTCACTAGGGAGTGCCAGACAGTGGGCGCAGGCCAGTGTGTGTGCGCACCGTGCGCGAGCCGAAGCAGGGCGAGGCATTGCCTCACCTGGGAAGCGCAAGGGGTCAGGGAGTTCCCTTTCCGAGTCAAAGAAAGGGGTGATGGACGCACCTGGAAAATCGGGTCACTCCCACCCGAATATTGCGCTTTTCAGACCGGCTTAAGAAACGGCGCACCACGAGACTATATCCCACACCTGGCTCAGAGGGTCCTACGCCCACGGAATC", "num_tokens": 102, # number of tokens to generate "top_k": 4, # only predict top 4 most likely outcomes per token "top_p": 1.0, # include 100% cumulative prob results in sampling "temperature": 0.7, # add variability (creativity) to predictions "": True, # enable more diverse outputs "enable_logits": False, # disable raw model output (logits)}# Retrieve the API key from the environmentkey = os.getenv("NVCF_RUN_KEY")# Send the example sequence and parameters to the Evo 2 APIr = requests.post( # Example URL for the Evo 2 model API. # Authorization headers to authenticate with the API headers={"Authorization": f"Bearer {key}"}, # The data payload (sequence and parameters) sent as JSON json=example,) |
有關各種提示的 API 輸出的更多信息,請參閱 NVIDIA BioNeMo 框架文檔 。
此外,還可以使用開源 NVIDIA BioNeMo Framework 對 Evo 2 進行微調,該框架提供可靠的工具,可根據 BioPharma 中的專業任務調整預訓練模型(例如 Evo 2):
# Prepare raw sequence data for training based on a YAML config file preprocess_evo2 -c data_preproc_config.yaml# Trains the Evo 2 model with preprocessed data and parallelism across multiple GPUs torchrun --nproc-per-node=8 --no-python train_Evo 2 -d data_train_config.yaml --num-nodes=1 --devices=8 --max-steps=100 --val-check-interval=25 --experiment-dir=/workspace/bionemo2/model/checkpoints/example --seq-length=8192 --tensor-parallel-size=4 --pipeline-model-parallel-size=1 --context-parallel-size=2 --sequence-parallel --global-batch-size=8 --micro-batch-size=1 --model-size=7b --fp8 --tflops-callback# Optional Fine-tuning: Add this argument to start from a pretrained model # --ckpt-dir=/path/to/pretrained_checkpoint |
Evo 2 和生物學領域的 AI 未來
AI 勢必會迅速改變生物學研究,實現之前人們認為需要數十年才能實現的突破。Evo 2 代表了這一變革的重大飛躍,它引入了基因組基礎模型,能夠以超大規模分析和生成 DNA、RNA 和蛋白質序列。
雖然 Evo 在預測原核生物的突變效應和基因表達方面表現優異,但 Evo 2 的功能更為廣泛,并增強了跨物種泛化,這使其成為研究真核生物學、人類疾病和進化關系的寶貴工具。
從識別導致癌癥風險的基因到設計復雜的生物分子系統,Evo 2 執行零樣本擊任務的能力充分體現了其通用性。借助長上下文依賴項,AI 能夠揭示跨基因組的模式,提供對精準醫學、農業和合成生物學的進步至關重要的多模態和多尺度見解。
隨著該領域的發展,像 Evo 2 這樣的模型為 AI 解讀生命復雜性的未來奠定了基礎,同時還用于設計新的有用的生物系統。這些進步與 AI 驅動的科學領域的更廣泛趨勢是一致的,在這些趨勢中,基礎模型針對特定領域的挑戰進行了定制,解鎖了以前無法實現的功能。Evo 2 的貢獻標志著 AI 將成為解碼、設計和重塑生存世界不可或缺的合作伙伴。
有關 Evo 2 的更多信息,請參閱 Arc Institute 發布的技術報告。Evo 2 也可在 NVIDIA BioNeMo 平臺中使用。
致謝?
在此, 我們要感謝以下參與所述研究的人員,感謝他們為本文的構思、寫作和圖形設計做出的杰出貢獻 :
- Garyk Brixi?,斯坦福大學遺傳學博士生
- 與 Arc Institute 合作的機器學習工程師 Jerome Ku
- Michael Poli?,Liquid AI 的創始科學家兼斯坦福大學計算機科學博士生
- Greg Brockman?,OpenAI 聯合創始人兼總裁
- Eric Nguyen,斯坦福大學生物工程博士生
- Brandon Yang,Cartesia AI 聯合創始人兼斯坦福大學計算機科學博士生 (休假中)
- Dave Burke,Arc Institute 首席技術官
- Hani Goodarzi?,Arc Institute 核心研究員,加州大學舊金山分校生物物理學和生物化學副教授
- Patrick Hsu?,Arc Institute 聯合創始人、生物工程助理教授兼加州大學伯克利分校 Deb 教職研究員
- Brian Hie?– 斯坦福大學化學工程助理教授、Dieter Schwarz 基金會斯坦福大學數據科學教職人員、Arc Institute 創新研究員、斯坦福大學進化設計實驗室負責人
?