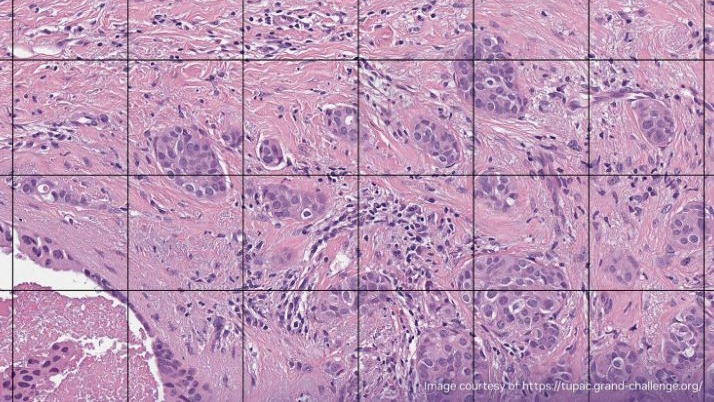
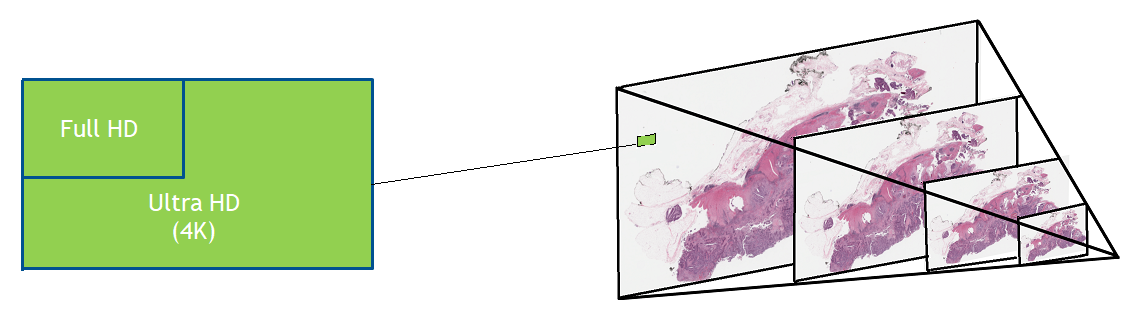
數字病理切片掃描儀生成大量圖像。載玻片通常以 40 倍的放大率進行掃描,得到千兆像素的圖像。壓縮可以將每個幻燈片的文件大小減少到 1 或 2 GB ,但這種數據量在移動、保存、加載和查看方面仍然具有挑戰性。要以全分辨率查看典型的完整幻燈片圖像,需要一個網球場大小的監視器。
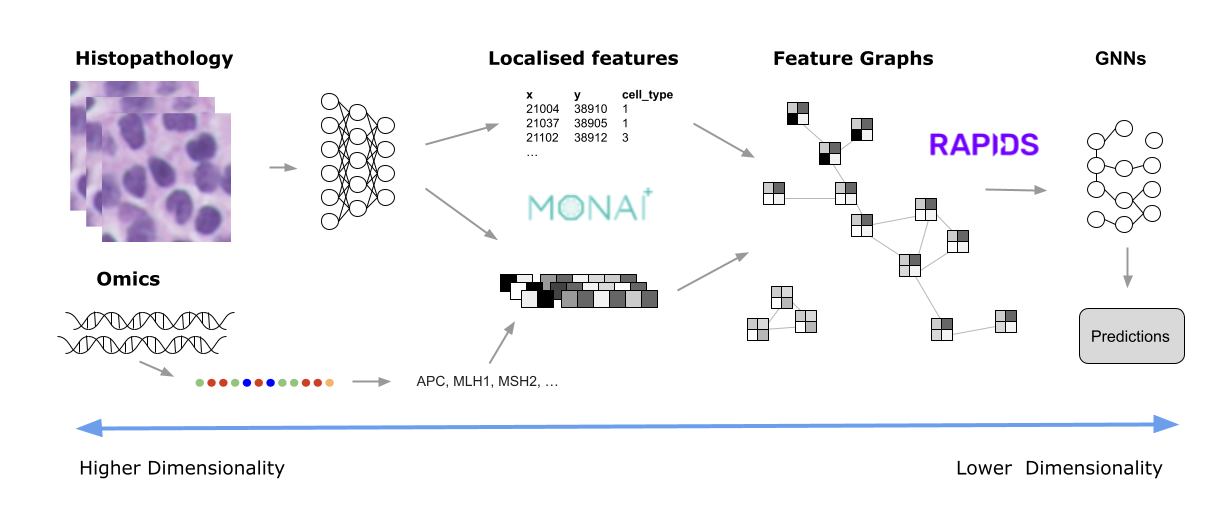
與組織病理學一樣,基因組學和顯微鏡可以產生數兆字節的數據。有些用例涉及多種模式,將這些數據轉換為更易于管理的大小通常需要進行漸進式轉換,直到只保留最顯著的特征。本文探討了實現這種數據細化的一些方法,使用的分析類型,以及諸如MONAI和RAPIDS可以釋放有意義的見解。以一個典型的數字組織病理學圖像為例,因為這些圖像現在在全球的常規臨床環境中使用。
MONAI 是一套開源、免費的協作框架,旨在加速醫學成像領域的研究和臨床協作。 RAPIDS 是一套開源軟件庫,用于在 GPU 上構建端到端的數據科學和分析管道。RAPIDS cuCIM 是一個用于多維圖像的計算機視覺處理軟件庫,可以加速 MONAI 的成像,以及cuDF library 可以幫助完成工作流所需的數據轉換。
管理整個幻燈片圖像數據
以前的研究表明cuCIM 可以加快整個幻燈片圖像的加載速度。例如,使用 cuCIM 加速 Scikit-Image API:在 GPU 上進行 n 維圖像處理和 I/O。
但是,管道的其余部分呢,可能包括圖像預處理、推理、后處理、可視化和分析?越來越多的儀器捕捉各種數據,包括多光譜圖像、遺傳和蛋白質組學數據,所有這些都面臨著類似的挑戰。
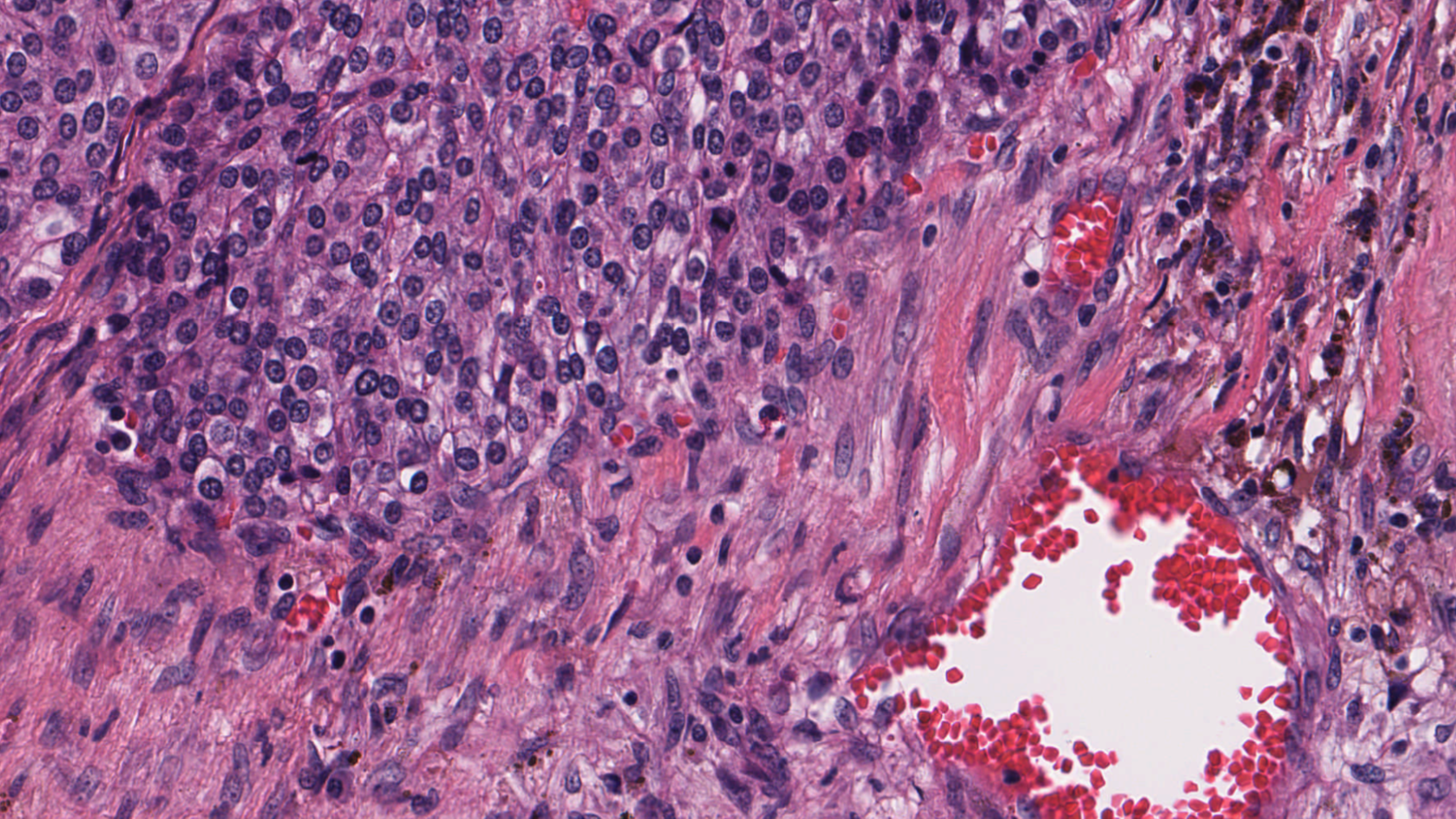
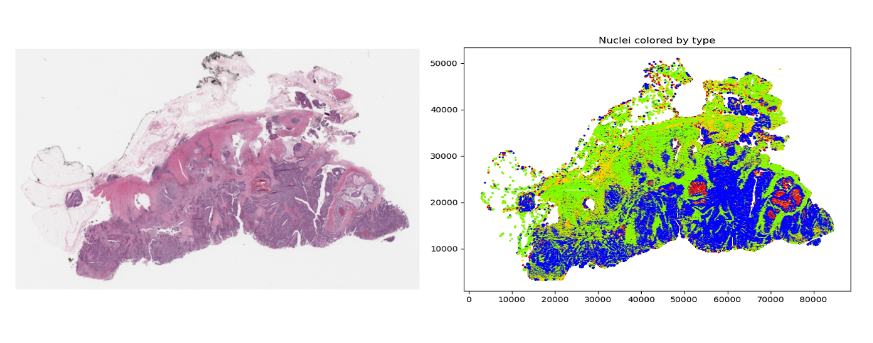
像癌癥這樣的疾病來自細胞核,細胞核只有約 5-20 微米大小。為了辨別各種細胞亞型,病理學家需要看到其形狀、顏色、內部紋理和模式。這需要非常大的圖像。
考慮到 2D 深度學習算法(如 DenseNet )的常見輸入大小通常約為 200 x 200 像素,高分辨率圖像只需一張幻燈片就需要分割成補丁——可能為 100000 個。
玻片制備和組織染色過程可能需要數小時。雖然低延遲推理結果的價值很小,但分析仍必須跟上數字掃描儀的采集率,以防止積壓。因此,吞吐量至關重要。提高吞吐量的方法是更快地處理圖像或同時計算多個圖像。
潛在解決方案
數據科學家和開發人員已經考慮了許多方法來使問題更容易處理。考慮到圖像的大小和病理學家進行診斷的時間有限,沒有實際的方法來以全分辨率查看每個像素。
相反,他們以較低的分辨率查看圖像,然后放大他們確定的可能包含感興趣特征的區域。他們通常可以在觀看了 1-2% 的全分辨率圖像后做出診斷。在某些方面,這就像犯罪現場的偵探:現場的大部分內容都無關緊要,結論通常取決于提供關鍵信息的一兩個纖維或指紋。
與人類同行不同,人工智能和機器學習( ML )無法丟棄圖像中 98-99% 的像素,因為擔心它們可能會錯過一些關鍵細節。這在未來可能是可能的,但需要相當大的信任和證據來證明它是安全的。
在這方面,?當前的算法對所有輸入像素一視同仁。各種算法機制隨后可以為它們分配或多或少的權重(注意力、最大池、偏差和權重),但最初它們都具有影響預測的相同潛力。
這不僅給組織病理學處理管道帶來了巨大的計算負擔,而且還需要在磁盤 CPU 和 GPU 之間移動大量數據。?大多數組織病理學幻燈片包含空白、冗余信息和噪音。可以利用這些特性來減少提取重要信息所需的實際計算。
例如,病理學家對相關區域內的某些細胞類型進行計數以對疾病進行分類可能就足夠了。要做到這一點,該算法必須將像素強度值轉換為具有相關細胞類型標簽的細胞核質心陣列。因此,計算一個區域內的細胞計數非常簡單。有許多方法可以將整個幻燈片圖像過濾為特定任務的基本元素。一些例子可能包括:
MONAI 和 RAPIDS
對于這些方法中的任一種,MONAI 提供了多種模型和訓練管道,您可以根據自己的需求進行自定義。大多數模型都是通用的,可以滿足數據的特定要求(如通道數量和維度),但也有一些是特定的,比如數字病理學。
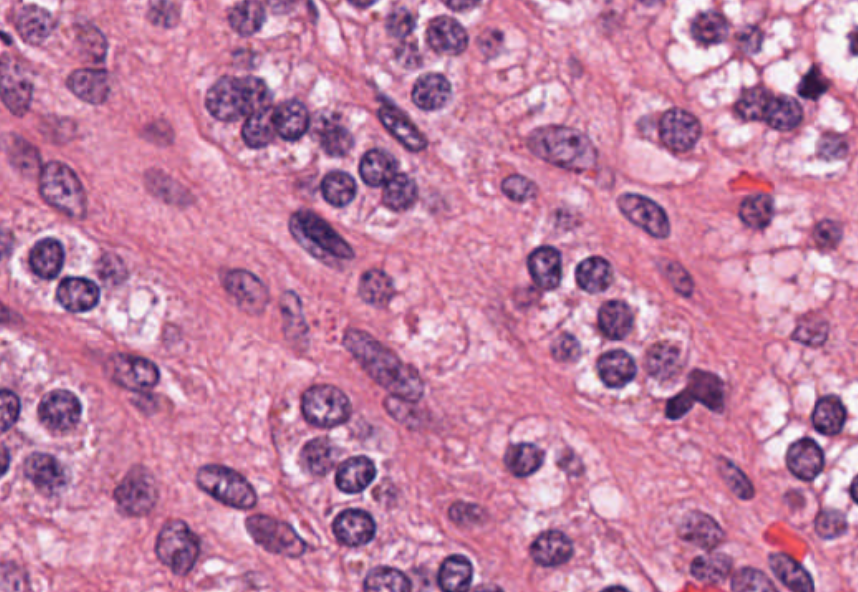
一旦導出了這些特征,就可以將其用于分析。然而,即使在這種類型的降維之后,仍可能有許多特征需要分析。例如,圖 3 顯示了一張包含數十萬個細胞核的圖像(最初為 100K x 60K RGB 像素)。即使為每個 64 x 64 瓦片生成嵌入,仍然可能為一張幻燈片生成數百萬個數據點。
這就是RAPIDS可以提供的幫助。它是一個開源 GPU 庫套件,使用 Python 加速數據科學,包括涵蓋一系列常見活動的工具,如機器學習(ML)、圖形分析、ETL 和可視化。有一些底層技術,例如CuPy,它使不同的操作能夠訪問 GPU 存儲器中的相同數據,而無需復制或重構底層數據。這是 RAPIDS 如此快速的主要原因之一。
開發人員的主要交互工具之一是 CUDA 加速的數據幀(cuDF)。數據以表格形式顯示,可以使用cuDF API,具有類似 pandas 的命令,易于采用。然后,這些數據幀被用作許多其他 RAPIDS 工具的輸入。
例如,假設您想從所有核創建一個圖,將每個核連接到某個半徑內的最近鄰居。為此,您需要向 cuGraph API 提供表示每個圖邊緣的源節點和目標節點的列(具有可選權重)。要生成此列表,可以使用 cuML 的最近鄰居搜索功能。只需提供一個列出所有核坐標的數據幀,cuML 就可以完成所有繁重的工作。
from cuml.neighbors import NearestNeighbors
knn = NearestNeighbors()
knn.fit(cdf)
distances, indices = knn.kneighbors(cdf, 5)請注意,默認情況下,計算的距離是歐幾里得距離,為了節省不必要的計算,它們是平方值。其次,該算法可以默認使用啟發式。如果需要實際值,可以指定可選的algorithm=‘brute’參數無論哪種方式,在 GPU 上的計算都非常快。
接下來,將距離和索引數據幀合并為一個單獨的數據幀。為此,需要首先為距離列指定唯一的名稱:
distances.columns=['ix2','d1','d2','d3','d4']
all_cols = cudf.concat(
[indices[[1,2,3,4]], distances[['d1','d2','d3','d4']]],
axis=1)
每一行都必須對應于圖中的一條邊,因此需要將數據幀拆分為每個最近鄰居的一行。然后,這些列可以重命名為“ source ”、“ target ”和“ distance ”
all_cols['index1'] = all_cols.index
c1 = all_cols[['index1',1,'d1']]
c1.columns=['source','target','distance']
c2 = all_cols[['index1',2,'d2']]
c2.columns=['source','target','distance']
c3 = all_cols[['index1',3,'d3']]
c3.columns=['source','target','distance']
c4 = all_cols[['index1',4,'d4']]
c4.columns=['source','target','distance']
edges = cudf.concat([c1,c2,c3,c4])
edges = edges.reset_index()
edges = edges[['source','target','distance']]消除所有?超過一定距離的鄰居,請使用以下過濾器:
distance_threshold = 15
edges = edges.loc[edges["distance"] < distance_threshold**2]在這一點上,您可以省去“距離”列,除非圖中的邊需要加權。然后創建圖形本身:
cell_graph = cugraph.Graph()
cell_graph.from_cudf_edgelist(edges,source='source', destination='target', edge_attr='distance', renumber=True)
有了圖形之后,就可以進行標準的圖形分析操作了。三角形計數是長度為三的循環數。圖的 k 核是包含 k 度或更高階節點的極大子圖:
count = cugraph.triangle_count(cell_graph)
coreno = cugraph.core_number(cell_graph)
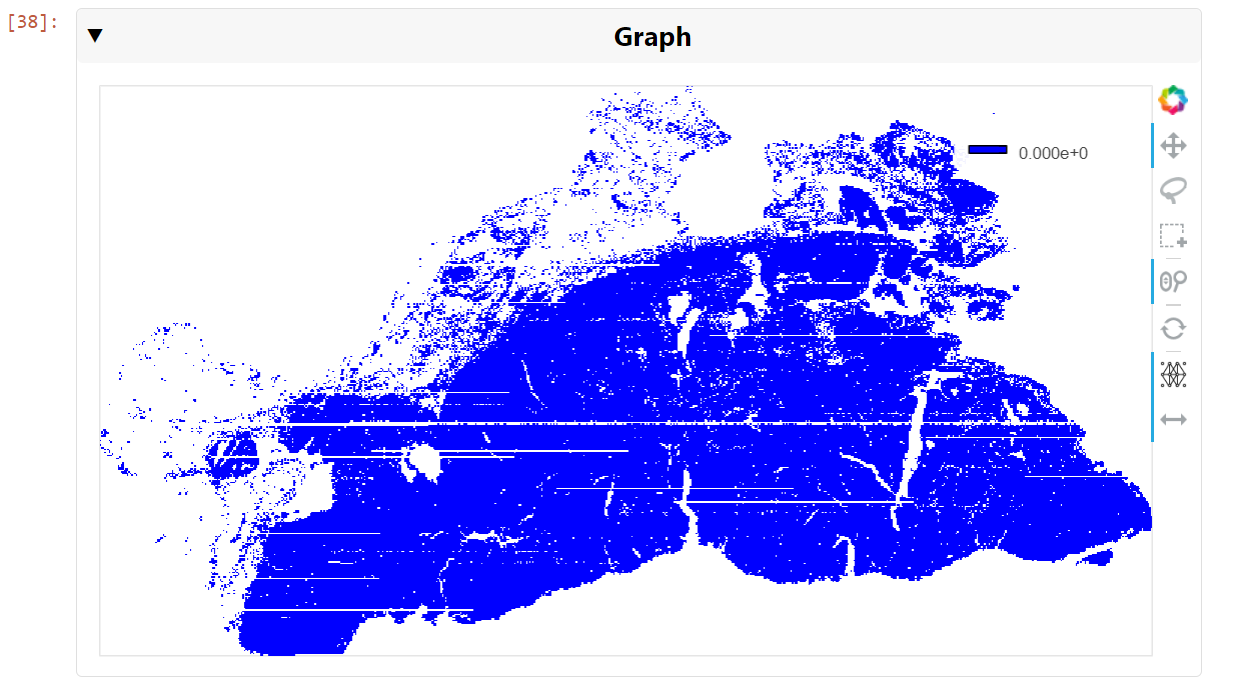
也可以可視化圖形,即使它可能包含數十萬條邊。使用現代 GPU,可以實時查看和導航圖形。要生成這樣的可視化效果,請使用 cuXFilter:
nodes = tiles_xy_cdf
nodes['vertex']=nodes.index
nodes.columns=['x','y','vertex']
cux_df = fdf.load_graph((nodes, edge_df))
chart0 = cfc.graph(
edge_color_palette=['gray', 'black'],
timeout=200,
node_aggregate_fn='mean',
node_pixel_shade_type='linear',
edge_render_type='direct',#other option available -> 'curved', edge_transparency=0.5)
d = cux_df.dashboard([chart0], layout=clo.double_feature)
chart0.view()
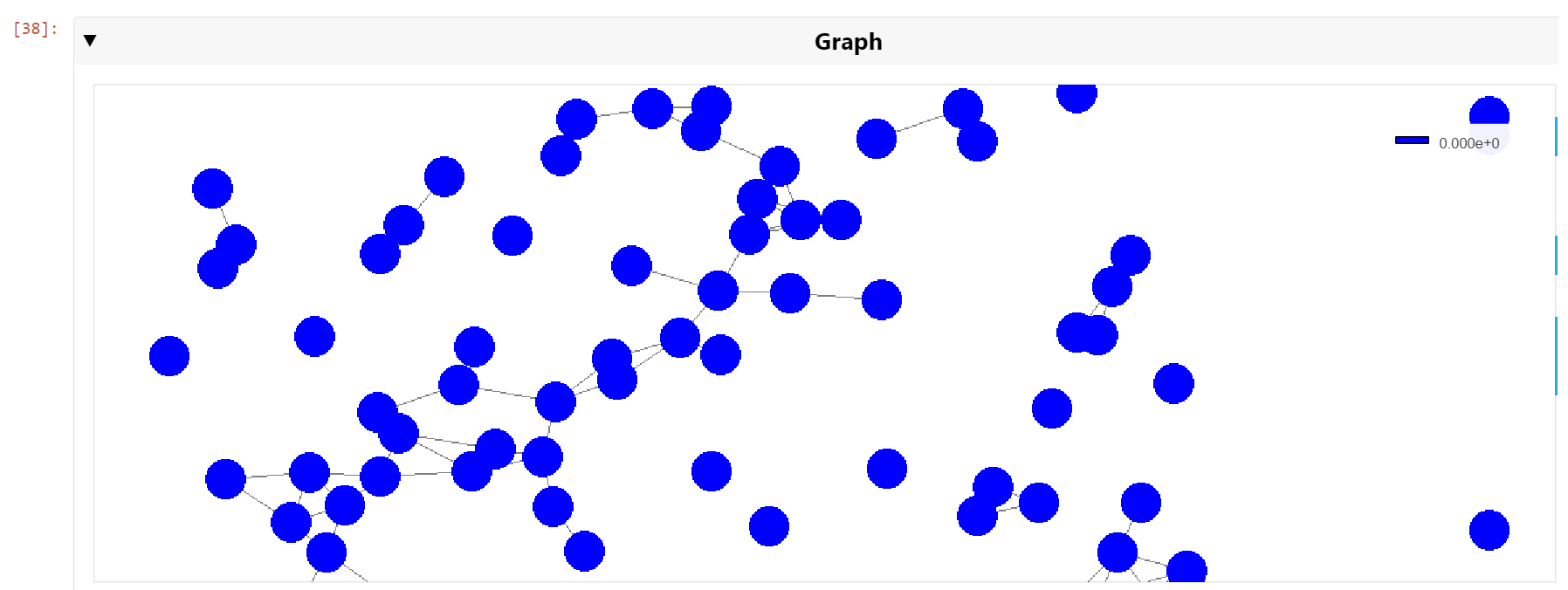
然后,您可以平移并縮小到細胞核級別,以查看最近鄰居的集群(圖 6 )。
結論
從原始像素中提取見解可能既困難又耗時。一些強大的工具和技術可以應用于大型圖像問題,即使是最具挑戰性的數據也可以提供近乎實時的分析。除了機器學習功能,GPU-RAPIDS等加速工具也提供了強大的可視化功能,有助于解讀基于深度學習的方法產生的計算特征。本文描述了一套端到端的工具,可以使用深度學習、機器學習圖和圖神經網絡方法。
使用RAPIDS和MONAI,在您的數據上釋放 GPU 的力量。加入MONAI 社區,在 NVIDIA 開發者論壇上參與討論。
?