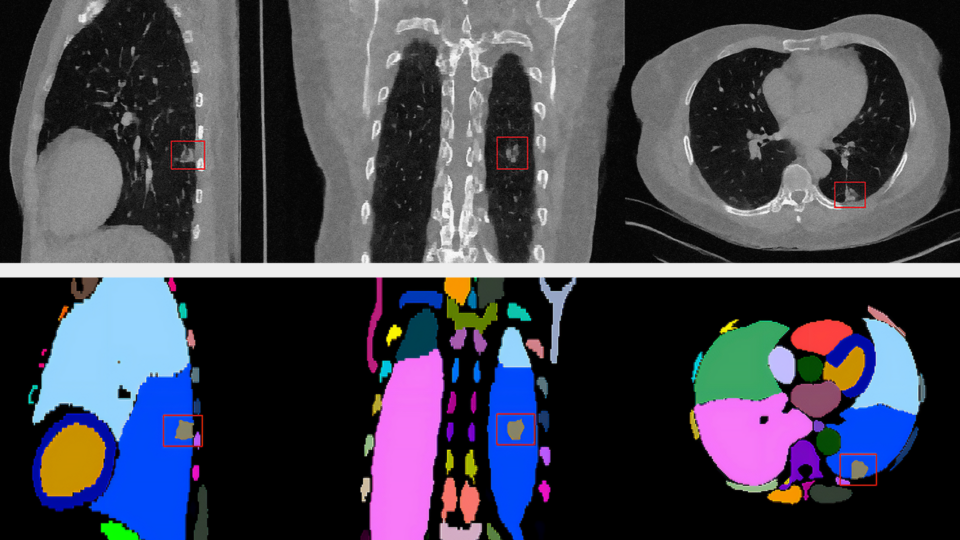
全球執行的計算機斷層掃描 (CT) 掃描次數超過 300M 次,僅在美國就有 85M 次。放射科醫生正在尋找加快工作流程并生成準確報告的方法,因此擁有一個基礎模型來分割所有器官和疾病會很有幫助。理想情況下,您可以通過優化的方式在生產中大規模運行此模型。
NVIDIA Research 創建了一個新的基礎模型,用于分割全身 CT 圖像,并將其打包到高度優化的容器中,以便在部署中進行擴展。在本文中,我們將討論 NVIDIA VISTA-3D 基礎模型, NVIDIA NIM,以及如何在數據上運行模型。
VISTA-3D
視覺基礎模型的興趣與日俱增。在醫學影像分析領域,兩個基本特征使這些模型特別實用:
- 對常見任務進行快速精確的推理
- 有效適應或零-shot 能力,以適應新任務
NVIDIA 一直專注于 3D CT 分割,最近開發了?VISTA-3D (Versatile Imaging SegmenTation and Annotation)。該模型已在超過 12,000 個體積上進行系統訓練,涵蓋 127 種人類解剖結構和各種病灶(肺結節、肝臟腫瘤、胰腺腫瘤、結腸腫瘤、骨病灶和腎腫瘤)。
它提供了精確的開箱即用分割,以及 state-of-the-art 的零樣本交互式分割。新的模型設計和訓練 recipe 代表了朝著開發多功能醫學影像基礎模型邁出的有希望的一步。
VISTA-3D 是一種領域專用的交互式基礎模型,它將語義分割與交互性相結合,為醫學成像的不同解剖學領域提供高精度和適應性。它具有以下核心工作流程:
- 分割一切:啟用全身探索,這對于了解影響多個器官的復雜疾病和整體治療規劃至關重要。
- 使用類別的分割:根據特定類別提供詳細的分割視圖,對于目標疾病分析或器官映射,例如關鍵器官中的腫瘤識別。
- 片段點提示:?通過用戶指導的基于單擊的選擇,提高了醫療影像分析中準確的真值數據創建的精度。這一種交互式方法加速了準確的真值數據的創建,這在醫學影像分析中至關重要。
圖 1 顯示了 VISTA-3D 架構的高級圖表,其中一個編碼器層后跟兩個并行解碼器層。一個解碼器用于自動分割,而另一個解碼器用于點提示。
每個頭部都會接收相應的輸入作為類提示或點點擊,由用戶指導分割。
每個頭部還會生成自己的分割結果,然后使用合并算法將這些結果合并到最終分割結果中。
有關該架構的更多信息,請參閱VISTA3D: Versatile Imaging Segmentation and Annotation model for 3D Computed Tomography研究論文。
VISTA-3D NIM 微服務
所有 NVIDIA NIM 微服務均托管在 NVIDIA API 目錄 上,以便您測試不同微服務并了解其功能。
在 Healthcare 下找到 VISTA-3D,并使用樣本數據對其進行測試。以 axial、coronal 或 sagittal 視圖查看測試數據集。VISTA-3D 可以分割超過 100 個器官,也可以選擇特定類別感興趣的器官。
使用 NVIDIA 托管的 NIM 微服務
您可以使用 NVIDIA 托管的 NIM 微服務對數據運行 VISTA-3D。注冊以獲取個人密鑰獲取個人密鑰。NVIDIA 為用戶提供 1000 個免費信用來試用任何 NIM 微服務。
為您的個人密鑰指定名稱和到期日期。在隨附的服務中包含 AI Foundation Models 和 Endpoints。您可以在所有 API 調用中使用此密鑰。有關更多信息,請參閱 Optionally 生成 NGC 密鑰。
要查看您的積分,請登錄到NVIDIA API Catalog并查看您的個人資料。您的積分列在右上角。
要測試首次 VISTA-3D NIM 微服務調用,請復制您喜歡的語言(shell 腳本、Python 或 Node.JS)的代碼,并使用最近生成的 API_KEY 值對您在 NVIDIA AI 解決方案頁面上使用的同一示例運行推理,類似于以下 Python 示例中所示。
import requests headers = {"Authorization": "Bearer <place you key here it should start with nvapi-> ",} filename="example-1"payload = { "output": {"extension": ".nii.gz","dtype": "uint8"}} session = requests.Session() response = session.post(invoke_url, headers=headers, json=payload) response.raise_for_status() file_name=f"{filename}_seg.nii.gz"with open(file_name, "wb") as file: file.write(response.content) print("File downloaded successfully!") |
恭喜,您剛剛對樣本數據運行了第一次 VISTA-3D NIM 微服務調用。這還確認了您的 API_KEY 值是否有效,并且您有足夠的 credits,現在您可以使用自己的數據進行測試。
每次推理調用都使用一個信用,您的信用在所有 NIM 微服務中共享。
使用數據運行 VISTA-3D
要在自己的數據上查看 VISTA-3D 推理,您必須設置一個 FTP 服務器來提供醫療圖像。
與接收可壓縮的小型文本負載的 LLM 模型不同,醫學影像通常很大。您無需在 API 負載中發送大型圖像,而是將圖像 URL 發送到 FTP 服務器。然后,VISTA-3D NIM 微服務從 FTP 服務器下載圖像并運行推理,然后再發送推理結果(Figure 2)。

在 GitHub 上共享文件
分享樣本數據的最簡單、最快捷的方法是使用 GitHub:
- 登錄您的 GitHub 帳戶。
- 創建新項目并確保它是公開的。
- 選擇“Upload file”(上傳文件),并上傳一些 NIfTI 文件,以 .nii 或 .nii.gz 為擴展名。確保每個文件的大小小于 25 MB。
- 選擇您的 NIfTI 文件,右鍵單擊Raw,然后選擇Copy link address。
生成的 URL 應該類似于以下示例:
https://github.com/<your_user_name>/Nifti_samples/raw/main/filename.nii.gz |
現在,您可以更改之前代碼示例中的 URL,以指向您的文件并進行下載。
payload = { "output": {"extension": ".nii.gz","dtype": "uint8"}} |
在云端共享樣本數據
如果云中已有一些數據,則可以公開少量 NIfTI 圖像以測試 VISTA-3D。在這種情況下,從本地文件系統目錄啟動簡單的 NGINX 服務器或簡單的 Python HTTP 服務器。這必須是公開可訪問的服務器或端口,以便 NVIDIA 托管的 VISTA-3D 微服務可以下載數據。
首先,使用 SSH 連接到服務器,移動一些示例數據,然后啟動簡單的 Python HTTP 文件服務器:
python -m http.server <port> |
您應該能夠使用 http://serverIP:port/ 從瀏覽器訪問這些文件。
連接到之前創建的 FTP 服務器,并更改行以指向您的 HTTP 服務器。
您會看到 HTTP 文件服務器響應了 NIM 請求,以下載圖像文件,然后您將獲得推理結果。
在本地運行 NIM 微服務
要開始在本地運行 NVIDIA NIM 微服務,請申請 NVIDIA NIM 訪問權限。申請完成后,NVIDIA 團隊將與您聯系以安排入門會議。在申請許可證之前,請務必等待指示,因為您需要 NVIDIA 許可證或試用許可證。
完成這些步驟并獲得批準后,您將獲得對 VISTA-3D NIM 微服務 Docker 容器的訪問權限。這使您能夠在本地或云端的首選硬件上運行微服務。
在以下部分中,我們將展示使用 Docker Compose 的示例設置,以幫助您快速啟動和運行。
預備知識
您應已安裝 Docker、Docker Compose 和 NVIDIA 驅動程序。要檢查,請運行以下命令,該命令提取最新 CUDA 版本的小鏡像,啟動 Docker 容器,然后檢查您是否可以從容器內訪問 GPU。
docker run --rm --gpus all nvidia/cuda: 12.5.0-base-ubuntu20.04 nvidia-smi |
您應該會看到一個輸出,其中顯示了系統上的 GPUs。
Docker Compose 文件
Docker Compose 代碼示例啟動兩個容器:
nim-vista運行推理nim-nginx來為您的圖像
# docker-compose.yml version: "3.9"services: nim: container_name: nim-vista image: nvcr.io/nvidia/nim/medical_imaging_vista3d:24.03 ports: - 8008:8008############################## nginx: container_name: nim-nginx image: nginx:1.19-alpine-perl volumes: - ./nginx.conf:/etc/nginx/nginx.conf - <local/folder/containing/nifti/files>:/files ports: - 8009:8009 |
將目錄更改為 local/folder/containing/nifti/files。
Nginx 基本配置
在同一文件夾中,創建nginx.conf 文件文件:
運行推理調用
要確認您的設置是否正確,請運行以下 curl 命令:
curl http://localhost:8008/health/ready |
此命令應返回 true,表明 VISTA-3D NIM 微服務已啟動并運行。
要確認nginx是否正確提供數據,請在瀏覽器上查看http://localhost:8009以查看您正在共享的文件夾和文件。
現在,您可以重復使用之前的相同代碼進行推理,只需稍作更改,即指向nginx以提供數據,而不是指向 NVIDIA 示例數據或您的 GitHub 上的示例文件。
import requests invoke_url = “http://localhost:8008/vista3d/inference” headers = {} filename="test_w_nginx"payload = { "output": {"extension": ".nii.gz","dtype": "uint8"}} session = requests.Session() response = session.post(invoke_url, headers=headers, json=payload) response.raise_for_status() file_name=f"{filename}_seg.nii.gz"with open(file_name, "wb") as file: file.write(response.content) print("File downloaded successfully!") |
現在,invoke_url 值指向您的主機,且 headers 為空,因為您無需使用 NGC 密鑰進行身份驗證。
運行后,您應該擁有包含所有標簽的 organs segmentation 文件。
安全提示
運行后,您可能不想讓 NGINX 端口打開,以便向任何有主機訪問權限的人提供數據。
改為限制對 VISTA-3D NIM 微服務的訪問。編輯 docker-compose.yml 文件,并刪除 ports: - 8009:8009 的行,以刪除 nginx 的端口映射。
接下來,將 infer 調用更改為使用nim-nginx而非localhost,后者已不可訪問。
"image":fhttps://nim-nginx:8809/{filename}.nii.gz |
當 Docker Compose 將所有服務置于同一網絡中時,NIM 可以訪問內部名稱 NIM-nginx。
結束語
在本文中,我們介紹了新的 NVIDIA AI 基礎模型 VISTA-3D,該模型可以在 CT 圖像中分割超過 100 個器官和多種疾病。我們還向您展示了 NVIDIA NIM 如何簡化使用這個模型的過程。
是否有興趣增強您的 CT 影像分析? 申請訪問 VISTA-3D NIM 微服務.獲得批準后,您可以在自己的硬件上運行此功能強大的模型,從而提高分割準確性并簡化工作流程。
?